Die ortsgerichtete Mutagenese (SDM) ist eine Technik, mit der eine oder mehrere Basen in einem Plasmid mutiert werden. Dieser Ansatz kann die Aminosäurenzusammensetzung verändern, Transkriptionsfaktor-Bindungsstellen zerstören oder Fusionsproteine erzeugen – um nur einige Beispiele zu nennen.
Obwohl die SDM am häufigsten zur Untersuchung der Struktur und biologischen Aktivität von Nukleinsäuren und Proteinen eingesetzt wird, ist sie auch nützlich, um Erkennungsstellen für Restriktionsenzyme einzuführen oder zu entfernen, um das Klonen zu erleichtern.
Außerdem kann die SDM ein nützliches Werkzeug zur Codon-Optimierung sein, um seltene Codons in Expressionsvektoren zu ersetzen, um die Effizienz der Expression heterologer Proteine zu verbessern. Dies ist manchmal aufgrund von Codon Bias notwendig, einem Phänomen, bei dem Organismen je nach Verfügbarkeit von tRNAs in der Zelle bestimmte Codons gegenüber anderen bevorzugen. Mehr über Codon Usage und Codon Bias erfahren Sie hier.
Wenn SDM eingesetzt wird, um die kodierende Sequenz zu verändern, entstehen spezifische Mutantenkonstrukte, denen kritische Reste oder Proteindomänen fehlen, die an posttranslationalen Modifikationen oder der Regulierung der Proteinstabilität beteiligt sind. Das Entfernen kritischer Reste oder Regionen in Ihren Plasmiden, wie z. B. Phosphorylierungsstellen oder kritische Domänen, bietet eine Möglichkeit, die Bedeutung bestimmter Proteinmerkmale zu demonstrieren. Die Veränderung von Phosphorylierungsstellen, z. B. von Serin zu nicht reaktivem Alanin oder zu Phosphomimetika wie Asparaginsäure, liefert zuverlässige Beweise für eine ortsspezifische Proteinphosphorylierung. Sie ermöglicht die Untersuchung der In-vitro-Implikationen solcher posttranslationalen Modifikationen.
Traditionelle Ansätze zur ortsgerichteten Mutagenese
Inverse PCR
Für Deletionen oder Insertionen von >50 bp ist die inverse PCR der beliebteste Ansatz. Bei der inversen PCR werden Back-to-Back-Primer verwendet, um das gesamte Plasmid zu amplifizieren, gefolgt von der Ligation des linearen Produkts unter Bildung zirkulärer DNA. Diese Technik eignet sich auch für größere Insertionen oder Deletionen, z. B. die Entfernung einer regulatorischen Domäne aus einem Protein.
Bei Deletionen kann der ausgewählte Bereich entfernt werden, indem Primer entworfen werden, die auf beiden Seiten der angestrebten Deletionszone annektieren. Die Amplifikation verläuft von diesem Bereich aus nach außen, so dass diese Region aus dem PCR-Produkt ausgeschlossen wird. Sobald das lineare Produkt ligiert ist, fehlt dem neuen Konstrukt diese Deletionsdomäne. Dieser Prozess ist in Abbildung 1 schematisch dargestellt.
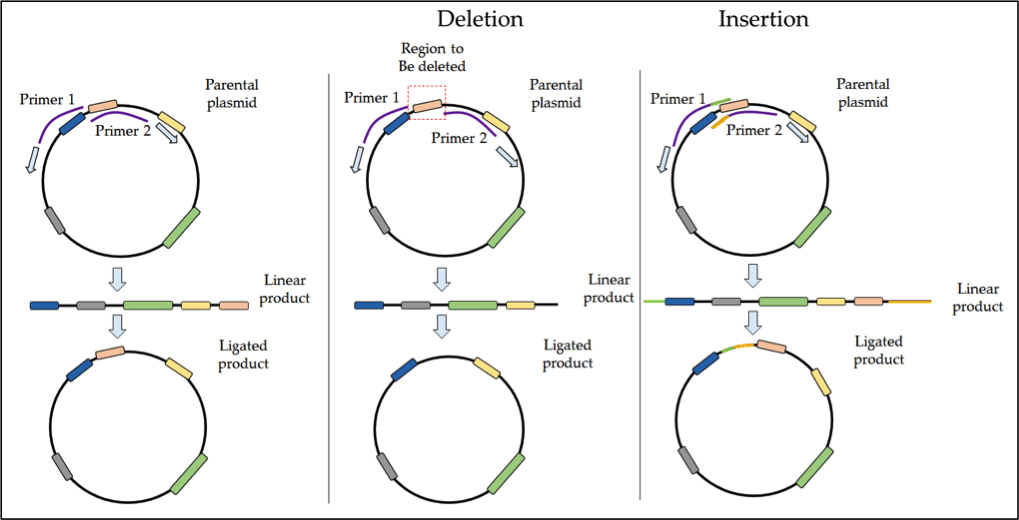
Abbildung 1. Protokoll für inverse PCR in SDM.
Insertionen können durch die Verwendung überlappender Primer mit flankierenden Sequenzen, die die entsprechenden zusätzlichen Basen enthalten, erreicht werden. Weitere Informationen über inverse PCR finden Sie in folgenden Quellen:
- Ochman et al. (1998) Genetic Applications of an Inverse Polymerase Chain Reaction. Genetics. 120:621-623.
- Clifford N. Dominy und David W. Andrews. Site-Directed Mutagenesis by Inverse PCR.
SDM Using Modified Primers
Diese Technik verwendet modifizierte Primer, um kleine Basenpaaränderungen in ein Plasmid einzubauen, und ist die Methode der Wahl für ortsspezifische Mutationen. Zu Beginn müssen Sie einige Primer entwerfen. Aber bevor Sie beginnen:
- Drucken Sie eine Kopie einer Aminosäure-Codon-Tabelle aus
- Halten Sie Ihre Plasmid- oder Proteinsequenz bereit.
- Stellen Sie sicher, dass Sie wissen, wo das Startcodon ist und in welchem Leseraster Ihre Sequenz gelesen wird.
Primerdesign
Suchen Sie nach SDM-Primern mit einer Länge von etwa 30 bp, wobei die mutierte Stelle so nah wie möglich an der Mitte liegen sollte. Es ist zwar akzeptabel, die Primer je nach Bedarf etwas länger oder kürzer zu machen, aber es sollten mindestens 12 bp auf jeder Seite der mutierten Stelle vorhanden sein.
Wenn Sie Hilfe beim Primerdesign benötigen, finden Sie in http://molbiol-tools.ca/PCR.htm einige sehr nützliche Ressourcen, die Ihnen den Weg weisen.
Wählen Sie die richtigen Reagenzien
Eine gewöhnliche Taq-Polymerase reicht bei SDM nicht aus – Sie müssen ein Korrekturenzym verwenden. Es gibt eine Reihe von kommerziell erhältlichen Polymerase-Kits, die für diese Aufgabe geeignet sind und eine Reihe von Merkmalen aufweisen, wie z. B. hohe Zuverlässigkeit (Korrekturlesefähigkeit) und Heißstartaktivierung. Eine Möglichkeit ist das In-Fusion HD Cloning Plus von Takara – eine Komplettlösung, die eine High-Fidelity-Polymerase, ein PCR-Reinigungskit, ein Klonierungsenzym und kompetente Zellen für die ortsgerichtete Mutagenese enthält.
Denken Sie daran, dass viele Polymerasen, wie die meisten Laborreagenzien, ihre eigenen Vor- und Nachteile haben – in der Regel haben die Labore historische Gründe für die Wahl der einen gegenüber der anderen und weichen selten davon ab. Auch wenn es vernünftig ist, bei dem zu bleiben, was funktioniert, kann es nicht schaden, die Literatur über andere verfügbare Ressourcen zu lesen, um sicherzustellen, dass Sie die besten Reagenzien für die Aufgabe haben!
PCR-Reaktion
Die verwendeten PCR-Bedingungen hängen von der Wahl des Kits und der Polymerase sowie von den Primern, die Sie entworfen haben, und von der Größe des Produkts ab. Das unten stehende Beispiel ist jedoch ein guter Ausgangspunkt.
| Schritt | Temp (°C) | Zeit (s) | Zyklen |
|---|---|---|---|
| Denatur | 94 | 15 | 18 |
| Anneal | 60 | 30 | 18 |
| Extension | 72 | 20/kb Plasmid | 18 |
Tabelle 1: Beispiel für ein SDM-Thermocycling-Programm
Wenn man die Anzahl der Zyklen niedrig hält, verhindert man das Auftreten unerwünschter Mutationen. Achtzehn Zyklen sollten eine angemessene Menge an mutiertem Produkt ergeben, ohne unerwünschte Mutationen einzubringen. Beachten Sie, dass die Anzahl der Zyklen während der Fehlersuche bei Bedarf geändert werden kann.
Digest It!
Nun, da Sie Ihr PCR-Produkt fertig haben, vergessen Sie nicht den entscheidenden Schritt – den Verdau! Hier verdauen Sie die elterliche Template-DNA, um sicherzustellen, dass Sie nur das mutierte Plasmid für die bakterielle Transformation haben. Standardprotokolle sehen eine einstündige Inkubation bei 37 °C mit der Endonuklease Dpn1 vor, um die gesamte dam- und hemi-methylierte elterliche DNA zu verdauen, so dass Sie das gewünschte mutierte Plasmid erhalten.
Transformieren und Sequenzieren
Transformieren Sie Ihr Plasmid in kompetente Zellen, wie Sie es mit jedem anderen Expressionsplasmid tun würden, und isolieren Sie einzelne Kolonien für die Plasmidisolierung.
Jetzt sollten Sie ein kleines Volumen Ihres gewünschten Plasmids haben. Bevor Sie mit dem Experimentieren beginnen, schicken Sie eine Probe zur Sequenzierung, um das Vorhandensein der gewünschten Modifikation(en) zu bestätigen. Genauso wichtig ist es, sicherzustellen, dass keine unerwünschten Sekundärmutationen vorhanden sind.
Wenn die Sequenzierung ein positives Ergebnis liefert – herzlichen Glückwunsch, Sie sind ein SDM-Profi! Aber wenn die Sequenzierung ein negatives Ergebnis liefert – kein Grund zur Panik! Sie können es immer wieder versuchen – unsere Expertentipps zur Fehlerbehebung bei SDM sollten Sie wieder auf den richtigen Weg bringen!
Die sinkenden Kosten für die Oligonukleotidsynthese und die Fortschritte in der synthetischen Biologie bedeuten, dass synthetische Ansätze gegenüber der ortsgerichteten Mutagenese immer mehr an Bedeutung gewinnen. Darüber hinaus hat das Aufkommen der CRISPR/Cas9-Technologie das Gene Editing vereinfacht, so dass die Mutagenese jetzt in vitro und in vivo in wenigen einfachen Schritten durchgeführt werden kann. Nichtsdestotrotz ist SDM weiterhin ein wichtiger Bestandteil des molekularbiologischen Werkzeugkastens und wird in absehbarer Zeit nicht verschwinden.
Ursprünglich veröffentlicht im Jahr 2016. Aktualisiert und neu veröffentlicht im Jahr 2018.

Hat Ihnen das geholfen? Dann teilen Sie es bitte mit Ihrem Netzwerk.
