La mutagénesis dirigida al sitio (SDM) es una técnica utilizada para mutar una o más bases dentro de un plásmido. Este enfoque puede cambiar la composición de aminoácidos, destruir los sitios de unión de los factores de transcripción o crear proteínas de fusión, por nombrar algunos ejemplos.
Aunque la SDM se utiliza más ampliamente para probar la estructura y la actividad biológica de los ácidos nucleicos y las proteínas, también es útil para introducir o eliminar los sitios de reconocimiento de las enzimas de restricción para ayudar a la clonación.
Además, la SDM puede ser una herramienta útil de optimización de codones para reemplazar los codones raros en los vectores de expresión para mejorar la eficiencia de la expresión de proteínas heterólogas. Esto es a veces necesario debido al sesgo de los codones, un fenómeno por el que los organismos tienen preferencia por ciertos codones sobre otros, dependiendo de la disponibilidad de ARNt en la célula. Puede leer más sobre el uso de codones y el sesgo de codones aquí.
Cuando se emplea para cambiar la secuencia codificante, el SDM crea construcciones mutantes específicas que carecen de residuos críticos o de dominios proteicos implicados en las modificaciones postraduccionales o en la regulación de la estabilidad de las proteínas. La eliminación de residuos o regiones críticas en sus plásmidos, como los sitios de fosforilación o los dominios críticos, proporciona una forma de demostrar la importancia de ciertas características de las proteínas. Por ejemplo, la alteración de los sitios de fosforilación, como la serina a una alanina no reactiva o a fosfomiméticos como el ácido aspártico, aporta pruebas fiables de la fosforilación de proteínas en un sitio específico. Permite el estudio de las implicaciones in vitro de dichas modificaciones postraduccionales.
Enfoques tradicionales de la mutagénesis dirigida al sitio
PCR inversa
Para deleciones o inserciones de >50 pb, la PCR inversa es el enfoque más popular. La PCR inversa utiliza cebadores consecutivos para amplificar todo el plásmido, seguido de la ligación del producto lineal formando ADN circular. Esta técnica también es adecuada para inserciones o deleciones más grandes, por ejemplo, para eliminar un dominio regulador de una proteína.
En el caso de las deleciones, la zona seleccionada puede eliminarse diseñando cebadores que se anillen a ambos lados de la zona de deleción objetivo. La amplificación procede hacia fuera de esta zona, excluyendo así esta región del producto de la PCR. Una vez ligado el producto lineal, su nueva construcción carecerá de este dominio de deleción. Este proceso se describe de forma esquemática en la Figura 1 a continuación.
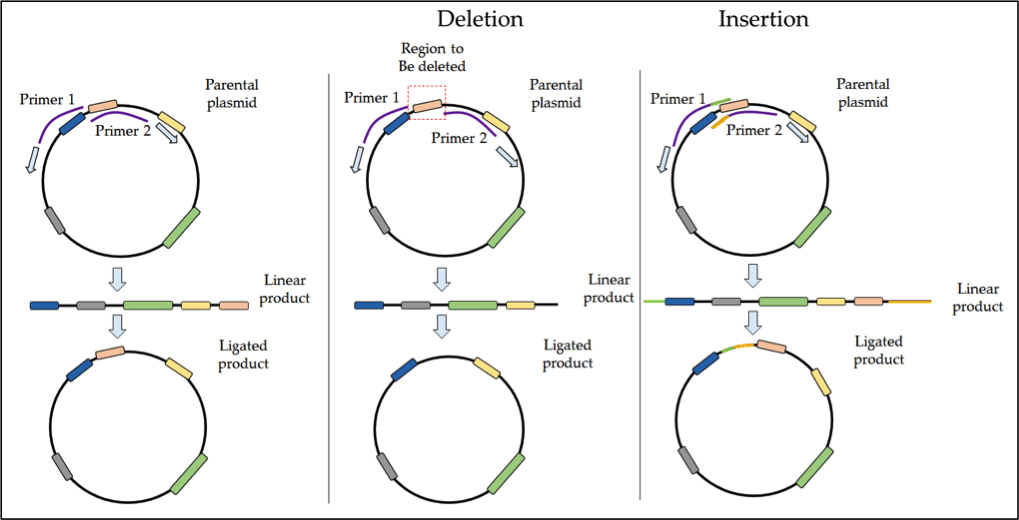
Figura 1. Protocolo para la PCR inversa en SDM.
Las inserciones pueden conseguirse utilizando cebadores superpuestos con secuencias de flanqueo que contengan las bases extra apropiadas. Para más información sobre la PCR inversa consulte estos recursos:
- Ochman et al. (1998) Genetic Applications of an Inverse Polymerase Chain Reaction. Genetics. 120:621-623.
- Clifford N. Dominy y David W. Andrews. Site-Directed Mutagenesis by Inverse PCR (Mutagénesis dirigida al lugar).
SDM Using Modified Primers
Esta técnica utiliza cebadores modificados para incorporar pequeños cambios de pares de bases en un plásmido, y es el método de elección para las mutaciones de sitio específico. Para empezar, necesitarás diseñar algunos cebadores. Pero antes de empezar:
- Imprima una copia de una tabla de codones de aminoácidos
- Tenga listo su plásmido o secuencia de proteínas.
- Asegúrese de saber dónde está el codón de inicio y en qué marco de lectura se lee su secuencia.
Diseño de los cebadores
Intente hacer cebadores SDM de aproximadamente 30 pb de longitud con su sitio mutado tan cerca del centro como sea posible. Aunque es aceptable hacer los cebadores un poco más largos o más cortos según sea necesario, debe haber un mínimo de 12 pb a cada lado de su sitio mutado.
Si necesita ayuda con el diseño del cebador, http://molbiol-tools.ca/PCR.htmenumera algunos recursos realmente útiles para ponerle en camino.
Elija los reactivos adecuados
La Taq polimerasa ordinaria no es suficiente cuando se trata de SDM – necesita usar una enzima correctora. Hay una variedad de kits de polimerasa disponibles en el mercado que están a la altura de la tarea, incorporando una serie de características que incluyen alta fidelidad (capacidad de corrección) y activación de arranque en caliente. Una posibilidad es la In-Fusion HD Cloning Plus de Takara, una solución todo en uno que incluye una polimerasa de alta fidelidad, un kit de purificación de PCR, una enzima de clonación y células competentes para la mutagénesis dirigida al sitio.
Tenga en cuenta que, al igual que la mayoría de los reactivos de laboratorio, muchas polimerasas tienen sus propias ventajas y desventajas. Aunque lo más sensato es quedarse con lo que funciona, no está de más leer la literatura de otros recursos disponibles para asegurarse de que tiene los mejores reactivos para el trabajo.
Reacción de PCR
Las condiciones de PCR utilizadas variarán dependiendo de su elección de kit y polimerasa, así como de los cebadores que haya diseñado y del tamaño del producto. Sin embargo, el ejemplo que se muestra a continuación es un buen punto de partida.
| Paso | Tem (°C) | Tiempo (s) | Ciclos |
|---|---|---|---|
| Denatura | 94 | 15 | 18 |
| Anneal | 60 | 30 | 18 |
| Extensión | 72 | 20/kb plásmido | 18 |
Tabla 1: Ejemplo de programa de ciclo térmico SDM
Mantener el número de ciclos bajo evitará que se produzcan mutaciones no deseadas. Dieciocho ciclos deberían producir una cantidad razonable de producto mutado sin incorporar mutaciones no deseadas. Tenga en cuenta que el número de ciclos puede modificarse durante la resolución de problemas si es necesario.
¡Digiéralo!
Ahora que tiene el producto de la PCR listo, no olvide el paso crítico: la digestión. Aquí, se digiere el ADN de la plantilla parental para asegurar que sólo se tiene el plásmido mutado para la transformación bacteriana. Los protocolos estándar exigen una incubación de 1 hora a 37°C con la endonucleasa Dpn1 para digerir todo el ADN parental dam-metilado y hemi-metilado, dejándolo con su plásmido mutado deseado.
Transformar y secuenciar
Transformar su plásmido en células competentes como lo haría con cualquier otro plásmido de expresión, y aislar colonias individuales para el aislamiento del plásmido.
Ahora debe tener un pequeño volumen de su plásmido deseado. Antes de comenzar a experimentar, envíe una muestra para la secuenciación a fin de confirmar la presencia de su(s) modificación(es) deseada(s). Es igualmente importante asegurar la ausencia de mutaciones secundarias no deseadas.
Si la secuenciación vuelve con un resultado positivo – ¡felicidades, eres un profesional del SDM! Pero si la secuenciación da un resultado negativo, ¡no se preocupe! Siempre puede volver a intentarlo: nuestros consejos de expertos para la resolución de problemas de SDM le permitirán volver a la pista. Además, la aparición de la tecnología CRISPR/Cas9 también ha simplificado la edición de genes, de modo que la mutagénesis puede realizarse ahora in vitro e in vivo en unos pocos y sencillos pasos. Sin embargo, el SDM sigue siendo un pilar en la caja de herramientas de la biología molecular, y no va a ir a ninguna parte pronto.
Publicado originalmente en 2016. Actualizado y republicado en 2018.

¿Te ha ayudado esto? Entonces por favor comparte con tu red.
