Nota del editor: Los artículos Platinum Highlight son publicaciones destacadas seleccionadas periódicamente por el Dr. Craig Reynolds, director asociado del Instituto Nacional del Cáncer, de entre las publicaciones Platinum más recientes.![]()
Cuando Alison Rattray y sus colegas del Laboratorio de Regulación Genética y Biología Cromosómica (GRCBL) examinaron una célula de levadura mutante que habían aislado en un cribado, observaron algo extraño.
El ADN mostraba una «reordenación muy específica, pero extraña», explicó. La disposición resultó ser un palíndromo de ADN, «abriendo la puerta al estudio de estos esquivos motivos de ADN», dijo.
Científica de plantilla en el GRCBL, Centro de Investigación del Cáncer del NCI, Rattray dijo que, aunque su descubrimiento se produjo hace varios años, su grupo sigue estudiando la reparación del ADN y los reordenamientos que dan lugar a una reparación anormal «debido a la asociación con ciertos cánceres.» El objetivo del grupo es comprender mejor el origen de estos reordenamientos, añadió.
Rattray es uno de los principales colaboradores de la investigación que se publica en BMC Genomics y que describe el método recientemente desarrollado por el grupo para la secuenciación de palíndromos de ADN en todo el genoma en una línea celular de cáncer.
¿Qué es un palíndromo de ADN?
Una secuencia palindrómica de nucleótidos (que se etiquetan como A, T, C o G) se produce cuando las cadenas complementarias de ADN se leen igual en ambas direcciones, ya sea desde el extremo 5-prima o desde el extremo 3-prima. Por ejemplo, la secuencia GGATCC en una hebra de ADN se considera un palíndromo porque la secuencia en su hebra complementaria es CCTAGG.
Debido a la complementariedad de las secuencias, las secuencias palindrómicas también pueden plegarse sobre sí mismas, formando bucles de horquilla o cruciformes que se extruyen de la doble hélice normal, dijo Rattray. «Las horquillas pequeñas no son problemáticas, pero cuando los palíndromos son largos (más de 100 pares de bases), interfieren en los procesos celulares normales, como la transcripción y la replicación», explicó.
Algunas células cancerosas presentan reordenamientos masivos del genoma, que incluyen amplificaciones, translocaciones y supresiones de genes, y estos reordenamientos suelen estar asociados a la presencia de un palíndromo, lo que sugiere una posible correlación entre el palíndromo y los reordenamientos génicos. No se sabe muy bien cómo surgen estos eventos. Lo que sí se sabe, sin embargo, es que tales reordenamientos se asocian con la progresión y el pronóstico del cáncer, dijo Rattray.
Nueva hipótesis sobre el reordenamiento genómico
Según Rattray, el modelo favorecido, propuesto originalmente hace más de 60 años por la doctora Barbara McClintock, sugiere que, tras una rotura cromosómica, las cromátidas hermanas se replican y fusionan, creando un cromosoma con dos centrómeros unidos por un palíndromo de ADN. En el modelo de McClintock, tener dos centrómeros da lugar a más reordenamientos genómicos.
Sin embargo, Rattray dijo que su grupo y otros han demostrado que «los palíndromos de ADN son inestables y pueden dar lugar a reordenamientos del genoma por sí mismos, lo que sugiere además que los palíndromos podrían surgir no sólo por la fusión de cromátidas hermanas, sino también por otros mecanismos, como los errores de replicación.»
El grupo planteó la hipótesis de que «en los cánceres que sufren reordenamientos masivos, las células son susceptibles de formar palíndromos, y una vez formados, la inestabilidad del palíndromo conduce a más reordenamientos, incluyendo la amplificación de genes, translocaciones y deleciones», dijo Rattray. «Cualquier reordenamiento de genes es mutagénico, y los reordenamientos que promueven el crecimiento celular, como en el cáncer, serán naturalmente favorecidos por la selección».
Una nueva tecnología identifica y caracteriza los palíndromos
Los investigadores desarrollaron una tecnología que les permitirá examinar los tumores, con el objetivo de comprender la probabilidad de que se formen palíndromos en ellos, dijo Rattray. Esperan saber qué acontecimientos inician estas formaciones inestables, y este nuevo conocimiento podría conducir a nuevos tratamientos. Por ejemplo, dijo, el grupo ya ha determinado que ciertas células de levadura susceptibles a la formación de palíndromos son mucho más sensibles que las células normales a la radiación, así como a los compuestos utilizados a menudo en el tratamiento del cáncer, como el cisplatino.
«Actualmente, estoy tratando de establecer métodos para enriquecer selectivamente los palíndromos del resto del ADN celular, lo que permitirá una mayor sensibilidad en el análisis del contenido de palíndromos de las células cancerosas», dijo. En el método anterior, los investigadores perdían las secuencias de unión que podían dar pistas sobre el origen de los palíndromos, y tenían que analizarlas una a una, explicó. «Ahora hemos demostrado que la plataforma PacBio puede secuenciar fácilmente a través de un palíndromo de ADN», dijo.
Rattray se doctoró en la Universidad de Washington, en Seattle, donde estudió la replicación retroviral. Tras una beca postdoctoral en la Universidad de Columbia, donde estudió la recombinación del ADN y los reordenamientos inducidos por roturas de doble cadena de ADN en la levadura, se incorporó al NCI en Frederick, trabajando en el laboratorio del doctor Jeffrey Strathern, jefe del GRCBL.
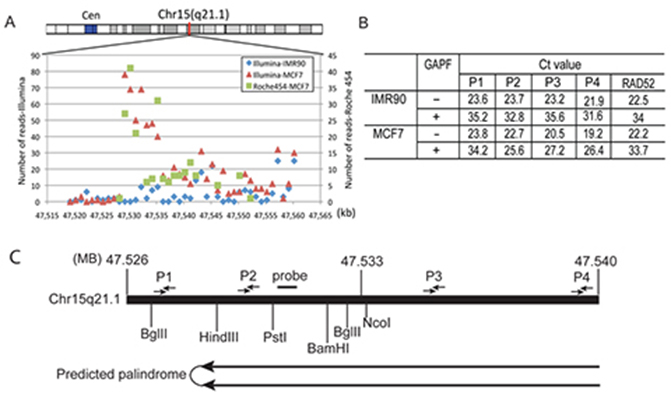
Estrategia de mapeo del palíndromo. (A) Distribución de la densidad de lectura en la Chr15q21.1: 47,529,204-47,550,373 región mostrada como bins de 1kb. (B) Análisis qPCR para controlar el enriquecimiento del palíndromo y determinar la direccionalidad del palíndromo Chr15q21.1. Los investigadores calcularon la cantidad de agotamiento de una región específica del conjunto de cebadores TaqMan basándose en el valor Ct antes y después del protocolo GAPF en las muestras IMR-90 y MCF-7. El enriquecimiento de pliegues se basa en la comparación del agotamiento de pliegues entre diferentes conjuntos de cebadores (P1, P2, P3 y P4) en relación con una secuencia de copia única en el genoma (RAD52). Las ubicaciones de los conjuntos de cebadores TaqMan P1, P2, P3 y P4 se indican en (C), mapa de la región genómica Chr15: 47.520.000-47.550.000 con los sitios de restricción y las ubicaciones de los cebadores. Figura de Yang et al., GAP-Seq: a method for identification of DNA palindromes, BMC Genomics 2014, 15:394; doi:10.1186/1471-2164-15-394.
.
