
La contribución de un puente salino a la estabilidad global al estado plegado de una proteína puede evaluarse a través de los datos termodinámicos recogidos en estudios de mutagénesis y técnicas de resonancia magnética nuclear. Utilizando una proteína de tipo pseudo-silvestre mutada específicamente para evitar la precipitación a un pH elevado, la contribución del puente salino a la energía libre global del estado plegado de la proteína puede determinarse realizando una mutación puntual, alterando y, en consecuencia, rompiendo el puente salino. Por ejemplo, se identificó la existencia de un puente salino en la lisozima T4 entre el ácido aspártico (Asp) en el residuo 70 y una histidina (His) en el residuo 31 (Figura 3). Se realizó una mutagénesis dirigida al sitio con asparagina (Asn) (Figura 4) obteniendo tres nuevos mutantes: Asp70Asn His31 (Mutante 1), Asp70 His31Asn (Mutante 2), y Asp70Asn His31Asn (Mutante doble).

Una vez establecidos los mutantes, pueden emplearse dos métodos para calcular la energía libre asociada a un puente salino. Un método implica la observación de la temperatura de fusión de la proteína de tipo salvaje frente a la de los tres mutantes. La desnaturalización puede controlarse mediante un cambio en el dicroísmo circular. Una reducción de la temperatura de fusión indica una reducción de la estabilidad. Esto se cuantifica a través de un método descrito por Becktel y Schellman en el que la diferencia de energía libre entre ambos se calcula a través de ΔTΔS. Este cálculo presenta algunos problemas y sólo puede utilizarse con datos muy precisos. En el ejemplo de la lisozima T4, la ΔS del tipo pseudo-suave se había notificado previamente a pH 5,5, por lo que la diferencia de temperatura del punto medio de 11 °C a este pH multiplicada por la ΔS notificada de 360 cal/(mol-K) (1,5 kJ/(mol-K)) arroja un cambio de energía libre de aproximadamente -4 kcal/mol (-17 kJ/mol). Este valor corresponde a la cantidad de energía libre aportada a la estabilidad de la proteína por el puente salino.
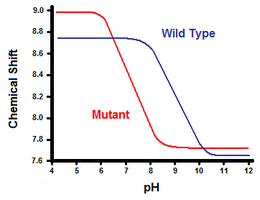
El segundo método utiliza la espectroscopia de resonancia magnética nuclear para calcular la energía libre del puente salino. Se realiza una valoración, mientras se registra el desplazamiento químico correspondiente a los protones del carbono adyacente al grupo carboxilato o amonio. El punto medio de la curva de valoración corresponde al pKa, o al pH en el que la proporción de moléculas protonadas:desprotonadas es de 1:1. Siguiendo con el ejemplo de la lisozima T4, se obtiene una curva de valoración mediante la observación de un desplazamiento del protón C2 de la histidina 31 (Figura 5). La figura 5 muestra el desplazamiento de la curva de valoración entre el tipo salvaje y el mutante en el que Asp70 es Asn. El puente salino formado es entre el Asp70 desprotonado y el His31 protonado. Esta interacción provoca el cambio observado en el pKa de His31. En la proteína desdoblada de tipo salvaje, en la que el puente salino está ausente, His31 tiene un pKa de 6,8 en tampones de H20 de fuerza iónica moderada. La figura 5 muestra un pKa del tipo salvaje de 9,05. Esta diferencia en el pKa se apoya en la interacción de His31 con Asp70. Para mantener el puente salino, His31 intentará mantener su protón el mayor tiempo posible. Cuando el puente salino se interrumpe, como en el mutante D70N, el pKa vuelve a un valor de 6,9, mucho más cercano al de His31 en el estado desplegado.
La diferencia en el pKa puede cuantificarse para reflejar la contribución del puente salino a la energía libre. Utilizando la energía libre de Gibbs:ΔG = -RT ln(Keq), donde R es la constante universal de los gases, T es la temperatura en kelvins, y Keq es la constante de equilibrio de una reacción en equilibrio. La desprotonación del His31 es una reacción de equilibrio ácido con una Keq especial conocida como la constante de disociación del ácido, Ka: His31-H+ ⇌ His31 + H+. El pKa se relaciona entonces con Ka mediante lo siguiente: pKa = -log(Ka). El cálculo de la diferencia de energía libre del mutante y del tipo salvaje puede hacerse ahora utilizando la ecuación de energía libre, la definición de pKa, los valores de pKa observados y la relación entre logaritmos naturales y logaritmos. En el ejemplo de la lisozima T4, este enfoque arrojó una contribución calculada de aproximadamente 3 kcal/mol a la energía libre global. Puede adoptarse un enfoque similar con el otro participante en el puente salino, como Asp70 en el ejemplo de la lisozima T4, controlando su cambio de pKa tras la mutación de His31.
Una palabra de precaución a la hora de elegir el experimento adecuado tiene que ver con la ubicación del puente salino dentro de la proteína. El entorno juega un papel importante en la interacción. A altas fuerzas iónicas, el puente salino puede quedar completamente enmascarado, ya que interviene una interacción electrostática. El puente salino His31-Asp70 en la lisozima T4 estaba enterrado dentro de la proteína. La entropía desempeña un papel más importante en los puentes salinos superficiales, en los que los residuos que normalmente tienen capacidad de movimiento están constreñidos por su interacción electrostática y los enlaces de hidrógeno. Se ha demostrado que esto disminuye la entropía lo suficiente como para casi borrar la contribución de la interacción. Los puentes salinos superficiales pueden estudiarse de forma similar a los puentes salinos enterrados, empleando ciclos de doble mutante y valoraciones de RMN. Aunque existen casos en los que los puentes salinos enterrados contribuyen a la estabilidad, como cualquier otra cosa, existen excepciones y los puentes salinos enterrados pueden mostrar un efecto desestabilizador. Asimismo, los puentes salinos superficiales, bajo ciertas condiciones, pueden mostrar un efecto estabilizador. El efecto estabilizador o desestabilizador debe evaluarse caso por caso y son pocas las afirmaciones generales que pueden hacerse.
