Szerkesztői megjegyzés: A Platinum Highlight cikkek olyan figyelemre méltó publikációk, amelyeket Dr. Craig Reynolds, a National Cancer Institute igazgatóhelyettese rendszeresen kiválaszt a legutóbb megjelent Platinum Publications közül.![]()
Amikor Alison Rattray és munkatársai a Génszabályozás és Kromoszómabiológiai Laboratóriumban (GRCBL) megvizsgáltak egy mutáns élesztősejtet, amelyet egy szűrés során izoláltak, valami furcsát vettek észre.
A DNS egy “nagyon specifikus, de furcsa átrendeződést” mutatott, magyarázta. Az elrendeződésről kiderült, hogy egy DNS-palindrom, ami “megnyitotta az ajtót az ilyen megfoghatatlan DNS-motívumok tanulmányozásához” – mondta.
A GRCBL, az NCI Rákkutatási Központjának munkatársa, Rattray elmondta, hogy bár felfedezésük már évekkel ezelőtt történt, csoportjuk továbbra is tanulmányozza a DNS-javítást és a rendellenes javítást eredményező átrendeződéseket, “mert bizonyos rákbetegségekkel kapcsolatban állnak”. A csoport célja, hogy jobban megértse ezeknek az átrendeződéseknek az eredetét – tette hozzá.
Rattray a BMC Genomics című folyóiratban közölt kutatás egyik fő szerzője, amely leírja a csoport által újonnan kifejlesztett módszert a DNS-palindrómák genomszintű szekvenálására egy rákos sejtvonalban.
Mi a DNS-palindrom?
A nukleotidok palindromos szekvenciája (amelyeket A, T, C vagy G-vel jelölnek) akkor fordul elő, amikor a DNS komplementer szálai mindkét irányban ugyanazt olvassák, akár az 5-prím végéről, akár a 3-prím végéről. Például a DNS egyik szálán lévő GGATCC szekvencia azért tekinthető palindromnak, mert a komplementer szálon lévő szekvencia CCTAGG.
A szekvencia komplementaritása miatt a palindromos szekvenciák magukra is visszahajolhatnak, hajszálhurkokat vagy feszületeket alkotva, amelyek a normál kettős spirálból kilépnek, mondta Rattray. “A kis hajtűkanyarok nem jelentenek problémát, de ha a palindromok hosszúak (több mint 100 bázispár), akkor zavarják a normális sejtfolyamatokat, például az átírást és a replikációt” – magyarázta.
Egyes rákos sejtek masszív genom-átrendeződéseket mutatnak, amelyek génamplikációkat, transzlokációkat és deléciókat tartalmaznak, és ezek az átrendeződések gyakran társulnak palindrom jelenlétével, ami a palindrom és a génátrendeződések közötti lehetséges összefüggésre utal. Az, hogy ezek az események hogyan keletkeznek, nem jól ismert. Azt azonban tudjuk, hogy az ilyen átrendeződések összefüggésben vannak a rák progressziójával és prognózisával, mondta Rattray.
Új hipotézis a genomiális átrendeződésről
Rattray szerint a kedvelt modell, amelyet eredetileg több mint 60 évvel ezelőtt Barbara McClintock, Ph.D. javasolt, azt sugallja, hogy egy kromoszómatörést követően a testvérkromatidák replikálódnak és összeolvadnak, létrehozva egy kromoszómát két centromerrel, amelyeket egy DNS-palindrom köt össze. A McClintock-modellben a két centroméra további genomi átrendeződésekhez vezet.
Rattray azonban elmondta, hogy az ő csoportja és mások kimutatták, hogy “a DNS-palindromok instabilak, és önmagukban is genomi átrendeződésekhez vezethetnek, ami arra utal, hogy a palindromok nem csak a testvérkromatidák fúziója, hanem más mechanizmusok, például replikációs hibák révén is létrejöhetnek”.”
A csoport feltételezte, hogy “a masszív átrendeződéseken áteső rákos megbetegedésekben a sejtek fogékonyak a palindromok kialakulására, és ha már kialakultak, a palindrom instabilitása további átrendeződésekhez vezet, beleértve a génamplifikációt, transzlokációkat és deléciókat” – mondta Rattray. “Minden génátrendeződés mutagén, és a sejtek növekedését elősegítő átrendeződéseket, mint a rákban, természetesen előnyben részesíti a szelekció.”
Új technológia azonosítja és jellemzi a palindromokat
A kutatók olyan technológiát fejlesztettek ki, amely lehetővé teszi számukra a daganatok vizsgálatát, azzal a céllal, hogy megértsék a palindromok kialakulásának valószínűségét ezekben a daganatokban, mondta Rattray. Remélik, hogy megtudják, milyen események indítják el ezeket az instabil képződményeket, és ez az új megértés újszerű kezelésekhez vezethet. Elmondta, hogy a csoport például már megállapította, hogy bizonyos élesztősejtek, amelyek hajlamosak a palindromok kialakulására, a normális sejteknél sokkal érzékenyebbek a sugárzásra, valamint a rák kezelésében gyakran használt vegyületekre, például a ciszplatinra.
“Jelenleg olyan módszereket próbálok kidolgozni, amelyekkel a palindromok szelektíven dúsíthatók a sejt DNS többi részéből, ami nagyobb érzékenységet tesz lehetővé a rákos sejtek palindromtartalmának elemzésében” – mondta. A korábbi módszerrel a kutatók elvesztették a csomóponti szekvenciákat, amelyek nyomokat adhatnak a palindromák eredetére, és egyenként kellett elemezniük őket – magyarázta. “Most megmutattuk, hogy a PacBio platform könnyedén képes szekvenálni egy DNS-palindromot” – mondta.”
Rattray a seattle-i Washington Egyetemen doktorált, ahol a retrovírusos replikációt tanulmányozta. A Columbia Egyetemen töltött posztdoktori ösztöndíját követően, ahol a DNS-rekombinációt és a DNS kettősszál-törések által indukált átrendeződéseket tanulmányozta élesztőben, a Frederick-i NCI-hez csatlakozott, ahol Dr. Jeffrey Strathern, a GRCBL vezetőjének laboratóriumában dolgozott.
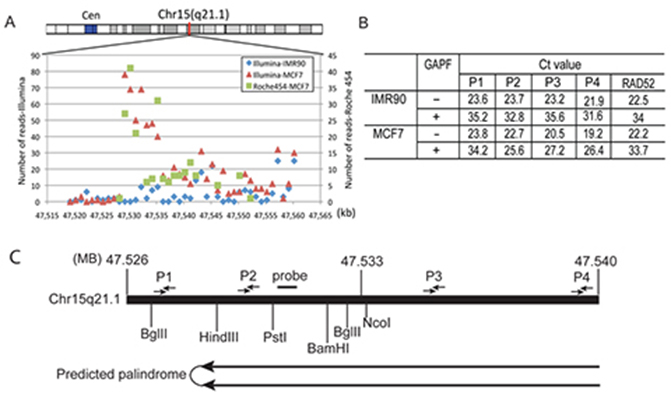
Palindrom-térképezési stratégia. (A) A leolvasási sűrűség eloszlása a Chr15q21.1-ben: 47,529,204-47,550,373 régió, 1 kb-os binsokként ábrázolva. (B) qPCR-analízis a palindrom-dúsulás monitorozására és a Chr15q21.1 palindrom irányultságának meghatározására. A kutatók a GAPF protokoll előtti és utáni Ct-érték alapján kiszámították egy adott TaqMan-primer készlet régió kiürülésének mértékét mind az IMR-90, mind az MCF-7 mintákban. A fold enrichment alapja a különböző primer szettek (P1, P2, P3 és P4) közötti depletio foldjának összehasonlítása a genomban található egyetlen másolatot tartalmazó szekvenciához (RAD52) viszonyítva. A P1, P2, P3 és P4 TaqMan-primer készletek helyét a (C), a Chr15: 47,520,000-47,550,000 genomi régió térképe a restrikciós helyekkel és a primerek helyével. Az ábra a Yang et al., GAP-Seq: a method for identification of DNA palindromes, BMC Genomics 2014, 15:394; doi:10.1186/1471-2164-15-394.
című cikkből származik.
