Nota redaktora: Artykuły Platinum Highlight to warte uwagi publikacje wybierane okresowo przez dr Craiga Reynoldsa, zastępcę dyrektora, National Cancer Institute, spośród ostatnio opublikowanych Platinum Publications.![]()
Gdy Alison Rattray i współpracownicy z Laboratorium Regulacji Genów i Biologii Chromosomów (GRCBL) zbadali zmutowaną komórkę drożdży, którą wyizolowali w ramach badań przesiewowych, zauważyli coś dziwnego.
DNA wykazywało „bardzo specyficzne, ale dziwne, rearanżacje”, wyjaśniła. Układ okazał się być palindromem DNA, „otwierając drzwi do badania tych nieuchwytnych motywów DNA”, powiedziała.
Pracownik naukowy w GRCBL, Centrum Badań nad Rakiem NCI, Rattray powiedział, że podczas gdy ich odkrycie miało miejsce kilka lat temu, ich grupa nadal bada naprawę DNA i rearanżacje, które powodują nieprawidłową naprawę „ze względu na związek z niektórymi nowotworami”. Celem grupy jest lepsze zrozumienie pochodzenia tych rearanżacji, dodała.
Rattray jest głównym współautorem badań opisanych w BMC Genomics, które opisują nowo opracowaną metodę grupy do sekwencjonowania genomu palindromów DNA w linii komórek nowotworowych.
Co to jest palindrom DNA?
Palindromiczna sekwencja nukleotydów (które są oznaczane jako A, T, C lub G) występuje wtedy, gdy komplementarne nici DNA czytane są tak samo w obu kierunkach, albo od końca 5-prime albo od końca 3-prime. Na przykład, sekwencja GGATCC na jednej nici DNA jest uważana za palindrom, ponieważ sekwencja na jego komplementarnej nici jest CCTAGG.
Z powodu komplementarności sekwencji, palindromiczne sekwencje mogą również złożyć z powrotem na siebie, tworząc pętle szpilki do włosów lub cruciforms, które są wyciągnięte z normalnej podwójnej helisy, Rattray powiedział. „Małe spinki do włosów nie są problematyczne, ale kiedy palindromy są długie (ponad 100 par zasad), zakłócają normalne procesy komórkowe, takie jak transkrypcja i replikacja,” wyjaśniła.
Niektóre komórki nowotworowe wykazują masywne rearanżacje genomu, które obejmują amplifikacje genów, translokacje i delecje, a te rearanżacje są często związane z obecnością palindromu, sugerując możliwy związek między palindromem a rearanżacjami genów. Jak powstają te zdarzenia nie jest dobrze poznane. Co jest znane, jednak, jest to, że takie rearanżacje są związane z progresją i rokowania raka, Rattray powiedział.
Nowa Hipoteza dla Genomic Rearrangement
Według Rattray, faworyzowany model, pierwotnie zaproponowany ponad 60 lat temu przez Barbara McClintock, Ph.D., sugeruje, że po przerwie chromosomu, siostrzane chromatydy replikują się i łączą, tworząc chromosom z dwoma centromerami połączonymi palindromem DNA. W modelu McClintocka, posiadanie dwóch centromerów prowadzi do dalszych rearanżacji genomowych.
Jednakże Rattray powiedziała, że jej grupa i inni pokazali, że „palindromy DNA są niestabilne i mogą prowadzić do rearanżacji genomu same w sobie, dalej sugerując, że palindromy mogą powstać nie tylko przez fuzję chromatyd siostrzanych, ale także przez inne mechanizmy, takie jak błędy replikacji.”Grupa postawiła hipotezę, że „w nowotworach, które przechodzą masywne rearanżacje, komórki są podatne na tworzenie palindromów, a gdy już powstaną, niestabilność palindromu prowadzi do dalszych rearanżacji, w tym amplifikacji genów, translokacji i delecji,” powiedział Rattray. „Wszelkie rearanżacje genów są mutagenne, a rearanżacje, które promują wzrost komórkowy, jak w raku, będą naturalnie faworyzowane przez selekcję”.
Nowa Technologia Identyfikuje i Charakteryzuje Palindromy
Badacze opracowali technologię, która umożliwi im badanie guzów, z celem zrozumienia prawdopodobieństwa powstawania palindromów w tych guzach, powiedział Rattray. Mają nadzieję dowiedzieć się, jakie wydarzenia inicjują takie niestabilne formacje, a to nowe zrozumienie może prowadzić do nowych metod leczenia. Na przykład, powiedziała, grupa już określiła, że niektóre komórki drożdży, które są podatne na tworzenie palindromów są znacznie bardziej wrażliwe niż normalne komórki na promieniowanie, jak również na związki często stosowane w leczeniu raka, takie jak cisplatin.
„Obecnie staram się ustanowić metody selektywnego wzbogacania palindromów z reszty komórkowego DNA, co pozwoli na większą czułość w analizie zawartości palindromów w komórkach nowotworowych,” powiedziała. W poprzedniej metodzie badacze tracili sekwencje łączące, które mogłyby dostarczyć wskazówek na temat pochodzenia palindromów, i musieli analizować je po kolei, wyjaśniła. „Teraz pokazaliśmy, że platforma PacBio może łatwo sekwencjonować przez palindrom DNA” powiedziała.
Rattray otrzymała doktorat na University of Washington, w Seattle, gdzie studiowała replikację retrowirusów. Po stażu podoktorskim na Uniwersytecie Columbia, gdzie badała rekombinację DNA i rearanżacje wywołane przez pęknięcia podwójnej nici DNA u drożdży, dołączyła do NCI w Frederick, pracując w laboratorium dr Jeffreya Stratherna, szefa GRCBL.
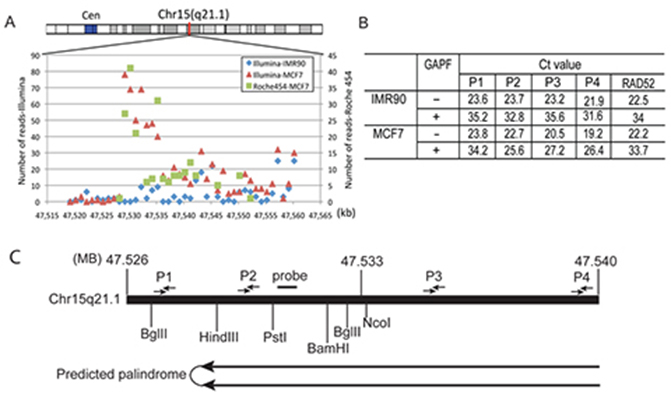
Strategia mapowania palindromów. (A) Rozkład gęstości odczytów w obszarze Chr15q21.1: 47,529,204-47,550,373 regionie przedstawionym jako 1-kb biny. (B) Analiza qPCR w celu monitorowania wzbogacania palindromów i określenia kierunkowości palindromu Chr15q21.1. Badacze obliczyli wielkość deplecji specyficznego regionu zestawu primerów TaqMan na podstawie wartości Ct przed i po protokole GAPF w próbkach IMR-90 i MCF-7. Fałdowe wzbogacenie oparte jest na porównaniu fałdowej deplecji pomiędzy różnymi zestawami primerów (P1, P2, P3 i P4) w stosunku do sekwencji o jednej kopii w genomie (RAD52). Lokalizacje zestawów primerów TaqMan P1, P2, P3 i P4 są wskazane w (C), mapa regionu genomu Chr15: 47,520,000-47,550,000 z miejscami restrykcyjnymi i lokalizacjami primerów. Rysunek za Yang i wsp., GAP-Seq: a method for identification of DNA palindromes, BMC Genomics 2014, 15:394; doi:10.1186/1471-2164-15-394.
.
