Mutageneza ukierunkowana miejscowo (SDM) jest techniką używaną do mutacji jednej lub więcej zasad w obrębie plazmidu. Podejście to może zmienić skład aminokwasów, zniszczyć miejsca wiązania czynników transkrypcyjnych lub stworzyć białka fuzyjne – to tylko kilka przykładów.
Mutageneza SDM jest najczęściej stosowana do badania struktury i aktywności biologicznej kwasów nukleinowych i białek, ale jest również przydatna do wprowadzania lub usuwania miejsc rozpoznawania enzymów restrykcyjnych w celu ułatwienia klonowania.
Co więcej, SDM może być użytecznym narzędziem optymalizacji kodonów w celu zastąpienia rzadkich kodonów w wektorach ekspresyjnych, aby poprawić wydajność ekspresji białek heterologicznych. Jest to czasami konieczne z powodu uprzedzenia kodonowego, zjawiska, w którym organizmy mają preferencje dla pewnych kodonów w stosunku do innych, w zależności od dostępności tRNA w komórce. Więcej o używaniu kodonów i tendencyjności kodonów można przeczytać tutaj.
Gdy stosuje się SDM do zmiany sekwencji kodującej, tworzy się specyficzne konstrukty mutantów, którym brakuje krytycznych reszt lub domen białkowych zaangażowanych w modyfikacje potranslacyjne lub regulację stabilności białka. Usuwanie krytycznych reszt lub regionów w plazmidach, takich jak miejsca fosforylacji lub krytyczne domeny, jest jednym ze sposobów na wykazanie znaczenia pewnych cech białka. Na przykład, zmiana miejsc fosforylacji, takich jak seryna do niereaktywnej alaniny lub do fosfomimetyków, takich jak kwas asparaginowy, dostarcza wiarygodnych dowodów na fosforylację białek specyficzną dla danego miejsca. Pozwala to na badanie in vitro implikacji takich modyfikacji potranslacyjnych.
Traditional Approaches to Site-Directed Mutagenesis
Inverse PCR
Dla delecji lub insercji >50 bp, inverse PCR jest najbardziej popularnym podejściem. Inverse PCR używa starterów back-to-back do amplifikacji całego plazmidu, po czym następuje ligacja liniowego produktu tworzącego kolisty DNA. Technika ta jest również odpowiednia dla większych insercji lub delecji, np. usunięcia domeny regulatorowej z białka.
Dla delecji, wybrany obszar może być usunięty poprzez zaprojektowanie primerów, które annealują po obu stronach docelowej strefy delecji. Amplifikacja przebiega na zewnątrz od tego obszaru, wykluczając w ten sposób ten region z produktu PCR. Po ligacji produktu liniowego, nowy konstrukt będzie pozbawiony domeny delecyjnej. Proces ten jest przedstawiony schematycznie na Rysunku 1 poniżej.
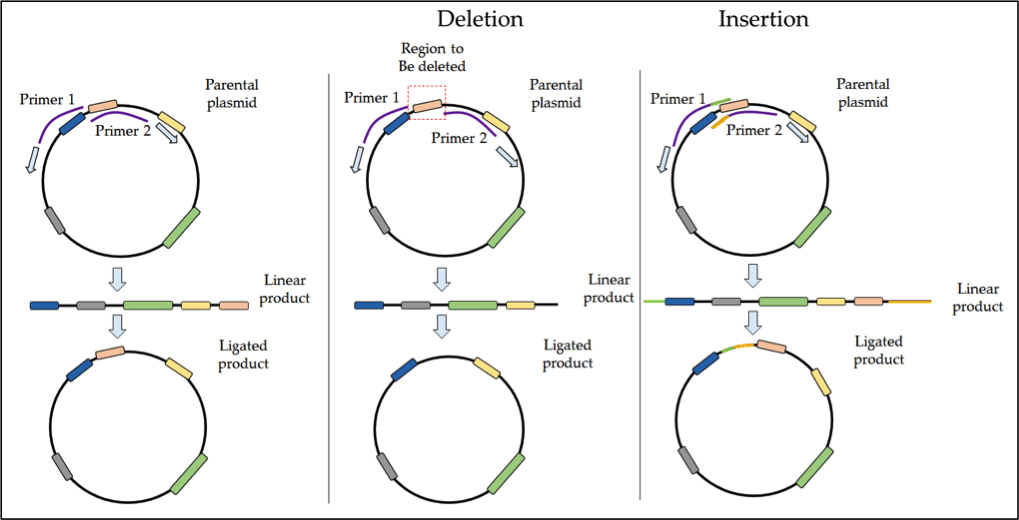
Rysunek 1. Protokół odwrotnego PCR w SDM.
Insercje można uzyskać stosując nakładające się startery z sekwencjami flankującymi, które zawierają odpowiednie dodatkowe zasady. Więcej informacji na temat odwróconej PCR można znaleźć w następujących źródłach:
- Ochman et al. (1998) Genetic Applications of an Inverse Polymerase Chain Reaction. Genetics. 120:621-623.
- Clifford N. Dominy and David W. Andrews. Site-Directed Mutagenesis by Inverse PCR.
SDM Using Modified Primers
Ta technika wykorzystuje zmodyfikowane startery do wprowadzenia małych zmian par zasad do plazmidu, i jest metodą z wyboru dla mutacji specyficznych dla miejsca. Na początek trzeba będzie zaprojektować kilka starterów. Ale zanim zaczniesz:
- Wydrukuj kopię tabeli kodonów aminokwasów
- Przygotuj swoją sekwencję plazmidu lub białka.
- Upewnij się, że wiesz, gdzie jest kodon startowy i w jakiej ramce odczytu jest odczytywana twoja sekwencja.
Projektowanie primerów
Dążenie do primerów SDM o długości około 30 bp z twoim zmutowanym miejscem tak blisko środka jak to możliwe. Chociaż dopuszczalne jest, aby startery były nieco dłuższe lub krótsze w zależności od potrzeb, powinno być minimum 12 bp po obu stronach Twojego zmutowanego miejsca.
Jeśli potrzebujesz pomocy w projektowaniu starterów, http://molbiol-tools.ca/PCR.htm wymienia kilka naprawdę użytecznych zasobów, aby ustawić Cię na swojej drodze.
Wybierz właściwe odczynniki
Zwykła polimeraza Taq po prostu nie będzie tego robić, jeśli chodzi o SDM – musisz użyć enzymu korekcyjnego. Istnieje wiele dostępnych na rynku zestawów polimerazy, które są odpowiednie do tego zadania i posiadają szereg cech, w tym wysoką wierność (zdolność do korekty) i aktywację gorącego startu. Jedną z możliwości jest In-Fusion HD Cloning Plus firmy Takara – rozwiązanie typu „wszystko w jednym”, które zawiera polimerazę o wysokiej wierności, zestaw do oczyszczania PCR, enzym do klonowania oraz komórki kompetentne do mutagenezy ukierunkowanej na miejsce.
Należy pamiętać, że podobnie jak większość odczynników laboratoryjnych, wiele polimeraz ma swoje wady i zalety – generalnie laboratoria będą miały historyczne powody, dla których wybierają jedną nad drugą i rzadko od tego odchodzą. Podczas gdy trzymanie się tego, co działa jest rozsądne, nie ma nic złego w czytaniu literatury na innych dostępnych źródłach, aby upewnić się, że masz najlepsze odczynniki do pracy!
Reakcja PCR
Użyte warunki PCR będą się różnić w zależności od wyboru zestawu i polimerazy, jak również starterów, które zaprojektowałeś i wielkości produktu. Jednakże, przykład pokazany poniżej jest dobrym punktem wyjścia.
| Krok | Temp (°C) | Czas (s) | Cykle |
|---|---|---|---|
| Denatura | 94 | 15 | 18 |
| Anneal | 60 | 30 | 18 |
| Extension | 72 | 20/kb plazmidu | 18 |
Tabela 1: Przykład programu termocyklingu SDM
Utrzymanie niskiej liczby cykli zapobiegnie wystąpieniu niepożądanych mutacji. Osiemnaście cykli powinno dać rozsądną ilość zmutowanego produktu bez włączania niepożądanych mutacji. Zauważ, że liczba cykli może być zmieniona podczas rozwiązywania problemów, jeśli jest to wymagane.
Trawić!
Teraz, gdy masz już gotowy swój produkt PCR, nie zapomnij o krytycznym kroku – trawieniu! Tutaj trawimy rodzicielski szablon DNA, aby upewnić się, że mamy tylko zmutowany plazmid do transformacji bakteryjnej. Standardowe protokoły wymagają 1-godzinnej inkubacji w 37°C z endonukleazą Dpn1, aby strawić całe dam-metylowane i hemi-metylowane rodzicielskie DNA, pozostawiając pożądany zmutowany plazmid.
Transformacja i sekwencjonowanie
Transformuj swój plazmid do komórek kompetentnych tak jak każdy inny plazmid ekspresyjny, i wyizoluj pojedyncze kolonie do izolacji plazmidu.
Teraz powinieneś mieć małą objętość swojego pożądanego plazmidu. Zanim zaczniesz eksperymentować, wyślij próbkę do sekwencjonowania, aby potwierdzić obecność pożądanej modyfikacji(y). Równie ważne jest zapewnienie braku jakichkolwiek wtórnych niepożądanych mutacji.
Jeśli sekwencjonowanie powróci z pozytywnym wynikiem – gratulacje, jesteś profesjonalistą SDM! Ale jeśli sekwencjonowanie zakończy się wynikiem negatywnym – nie martw się! Zawsze możesz spróbować ponownie – nasze wskazówki dla ekspertów dotyczące rozwiązywania problemów z SDM powinny przywrócić Cię na właściwą drogę!
W dzisiejszych czasach malejące koszty syntezy oligonukleotydów i postępy w biologii syntetycznej oznaczają, że podejścia syntetyczne zyskują na znaczeniu w stosunku do mutagenezy ukierunkowanej na miejsce. Ponadto, pojawienie się technologii CRISPR/Cas9 również uprościło edycję genów tak, że mutageneza może być obecnie przeprowadzana in vitro i in vivo w kilku prostych krokach. Niemniej jednak SDM nadal jest ostoją w zestawie narzędzi biologii molekularnej i nigdzie się nie wybiera w najbliższym czasie.
Originally published in 2016. Zaktualizowane i ponownie opublikowane w 2018 roku.

Czy to pomogło Ci? Następnie proszę podzielić się ze swoją siecią.
.
