Nota editorului: Articolele Platinum Highlight sunt publicații demne de luat în seamă, selectate periodic de Dr. Craig Reynolds, director asociat al Institutului Național de Cancer, dintre cele mai recente publicații Platinum publicate.![]()
Când Alison Rattray și colegii din Laboratorul de Reglementare Genetică și Biologie a Cromozomului (GRCBL) au examinat o celulă de drojdie mutantă pe care o izolaseră în cadrul unui screening, au observat ceva ciudat.
ADN-ul prezenta o „rearanjare foarte specifică, dar ciudată”, a explicat ea. Aranjamentul s-a dovedit a fi un palindrom de ADN, „deschizând ușa pentru a studia aceste motive evazive ale ADN-ului”, a spus ea.
Un cercetător științific din cadrul GRCBL, NCI Center for Cancer Research, Rattray a spus că, deși descoperirea lor a avut loc cu câțiva ani în urmă, grupul lor continuă să studieze repararea ADN-ului și rearanjamentele care au ca rezultat o reparare anormală „din cauza asocierii cu anumite tipuri de cancer”. Scopul grupului este de a înțelege mai bine originea acestor rearanjamente, a adăugat ea.
Rattray este unul dintre principalii contribuitori la cercetarea raportată în BMC Genomics care descrie metoda recent dezvoltată de grup pentru secvențierea la nivel de genom a palindromurilor ADN într-o linie celulară canceroasă.
Ce este un palindrom de ADN?
O secvență palindromică de nucleotide (care sunt etichetate A, T, C sau G) apare atunci când șirurile complementare de ADN se citesc la fel în ambele direcții, fie de la capătul de 5 prime, fie de la capătul de 3 prime. De exemplu, secvența GGATCC de pe o catenă de ADN este considerată un palindrom deoarece secvența de pe catena sa complementară este CCTAGG.
Din cauza complementarității secvențelor, secvențele palindromice se pot, de asemenea, plia pe ele însele, formând bucle în formă de ac de păr sau cruciforme care sunt extrudate din dublul helix normal, a spus Rattray. „Pinii de păr mici nu sunt problematici, dar atunci când palindromurile sunt lungi (mai mult de 100 de perechi de baze), acestea interferează cu procesele celulare normale, cum ar fi transcrierea și replicarea”, a explicat ea.
Câteva celule canceroase prezintă rearanjamente masive ale genomului, care includ amplificări, translocații și deleții de gene, iar aceste rearanjamente sunt adesea asociate cu prezența unui palindrom, sugerând o posibilă corelație între palindrom și rearanjamentele genice. Modul în care apar aceste evenimente nu este bine înțeles. Ceea ce se știe, însă, este că astfel de rearanjamente sunt asociate cu progresia și prognosticul cancerului, a spus Rattray.
Nouă ipoteză pentru rearanjarea genomică
Potrivit lui Rattray, modelul favorizat, propus inițial cu peste 60 de ani în urmă de Barbara McClintock, Ph.D., sugerează că, în urma unei rupturi cromozomiale, cromatidele surori se replică și fuzionează, creând un cromozom cu doi centromeri uniți de un palindrom de ADN. În modelul McClintock, faptul de a avea doi centromeri duce la rearanjamente genomice ulterioare.
Cu toate acestea, Rattray a spus că grupul său și alții au arătat că „palindromii de ADN sunt instabili și pot duce la rearanjamente genomice de la sine, sugerând în continuare că palindromii ar putea apărea nu numai prin fuziunea cromatidelor surori, ci și prin alte mecanisme, cum ar fi erorile de replicare.”
Grupul a emis ipoteza că „în cancerele care suferă rearanjamente masive, celulele sunt predispuse la formarea palindromurilor, iar odată formate, instabilitatea palindromului duce la alte rearanjamente, inclusiv la amplificarea genelor, translocații și deleții”, a spus Rattray. „Orice rearanjare genetică este mutagenă, iar rearanjamentele care promovează creșterea celulară, ca în cazul cancerului, vor fi favorizate în mod natural de selecție.”
Noua tehnologie identifică și caracterizează palindromurile
Cercetătorii au dezvoltat o tehnologie care le va permite să examineze tumorile, cu scopul de a înțelege probabilitatea formării palindromurilor în aceste tumori, a spus Rattray. Ei speră să afle ce evenimente inițiază astfel de formațiuni instabile, iar această nouă înțelegere ar putea duce la tratamente noi. De exemplu, a spus ea, grupul a determinat deja că anumite celule de drojdie care sunt susceptibile la formarea de palindromi sunt mult mai sensibile decât celulele normale la radiații, precum și la compușii utilizați adesea în tratamentul cancerului, cum ar fi cisplatinul.
„În prezent, încerc să stabilesc metode de îmbogățire selectivă a palindromilor din restul ADN-ului celular, ceea ce va permite o mai mare sensibilitate în analiza conținutului de palindromi din celulele canceroase”, a spus ea. În metoda anterioară, cercetătorii pierdeau secvențele de joncțiune care ar putea oferi indicii cu privire la originea palindromurilor și trebuiau să le analizeze unul câte unul, a explicat ea. „Am demonstrat acum că platforma PacBio poate secvenția cu ușurință printr-un palindrom de ADN”, a spus ea.
Rattray și-a obținut doctoratul la Universitatea din Washington, în Seattle, unde a studiat replicarea retrovirală. După o bursă postdoctorală la Universitatea Columbia, unde a studiat recombinarea ADN-ului și rearanjamentele induse de rupturi de ADN dublu catenar în drojdie, ea s-a alăturat NCI la Frederick, lucrând în laboratorul lui Jeffrey Strathern, Ph.D., șef, GRCBL.
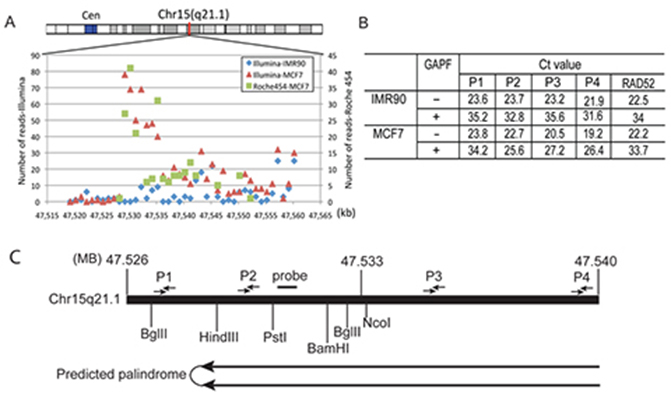
Strategia de cartografiere a palindromului. (A) Distribuția densității de citire în Chr15q21.1: 47,529,204-47,550,373 regiune reprezentată ca bini de 1 kb. (B) Analiza qPCR pentru a monitoriza îmbogățirea palindromului și pentru a determina direcționalitatea palindromului Chr15q21.1. Cercetătorii au calculat cantitatea de sărăcire a unei regiuni specifice din setul de amorsă TaqMan pe baza valorii Ct înainte și după protocolul GAPF atât în probele IMR-90, cât și în probele MCF-7. Îmbogățirea fold se bazează pe compararea depleției fold între diferite seturi de primer (P1, P2, P3 și P4) în raport cu o secvență cu o singură copie în genom (RAD52). Locațiile seturilor de amorsă TaqMan P1, P2, P3 și P4 sunt indicate în (C), harta regiunii genomice Chr15: 47,520,000-47,550,000 cu situsurile de restricție și locațiile amorselor. Figura din Yang et al., GAP-Seq: a method for identification of DNA palindromes, BMC Genomics 2014, 15:394; doi:10.1186/1471-2164-15-394.
.
