Toimittajan huomautus: Platinum Highlight -artikkelit ovat huomionarvoisia julkaisuja, jotka tohtori Craig Reynolds, apulaisjohtaja, National Cancer Institute, valitsee säännöllisin väliajoin viimeisimpien Platinum-julkaisujen joukosta.![]()
Kun Alison Rattray ja kollegansa geenien säätelyn ja kromosomibiologian laboratoriossa (Gene Regulation and Chromosome Biology Laboratory, GRCBL) tutkivat mutanttista hiivasolua, jonka he olivat eristäneet seulonnassa, he huomasivat jotakin outoa.
DNA:lla näkyi ”hyvin spesifinen, mutta omituinen uudelleenjärjestäytyminen”
Rattrayn mukaan. Järjestely osoittautui DNA-palindromiksi, mikä ”avasi oven näiden vaikeasti hahmotettavien DNA-motiivien tutkimiseen”, hän sanoi.
NCI:n syöpätutkimuskeskuksen GRCBL:ssä työskentelevä tutkija Rattray sanoi, että vaikka heidän löytönsä tapahtui useita vuosia sitten, heidän ryhmänsä jatkaa DNA:n korjauksen ja epänormaaliin korjaukseen johtavien uudelleenjärjestelyjen tutkimista, ”koska niillä on yhteys tiettyihin syöpiin”. Ryhmän tavoitteena on ymmärtää paremmin näiden uudelleenjärjestelyjen alkuperää, hän lisäsi.
Rattray on merkittävä tekijä BMC Genomics -lehdessä raportoidussa tutkimuksessa, jossa kuvataan ryhmän hiljattain kehittämä menetelmä DNA-palindromien genominlaajuiseen sekvensointiin syöpäsolulinjassa.
Mikä on DNA-palindromi?
Nukleotidien (jotka merkitään A:lla, T:llä, C:llä tai G:llä) palindrominen sekvenssi syntyy, kun DNA:n komplementaariset säikeet lukevat samoja molempiin suuntiin, joko 5-päästä tai 3-päästä. Esimerkiksi yhdellä DNA-juosteella olevaa sekvenssiä GGATCC pidetään palindromina, koska sen komplementaarisen juosteen sekvenssi on CCTAGG.
Sekvenssin komplementaarisuuden vuoksi palindromiset sekvenssit voivat myös taittua takaisin itseensä muodostaen hiusneulasilmukoita tai ristikoita, jotka puristuvat ulos normaalista kaksoiskierteestä, Rattray sanoi. ”Pienet hiuslenkit eivät ole ongelmallisia, mutta kun palindromit ovat pitkiä (yli 100 emäsparia), ne häiritsevät normaaleja soluprosesseja, kuten transkriptiota ja replikaatiota”, hän selitti.
Joissakin syöpäsoluissa esiintyy massiivisia perimän uudelleenjärjestelyjä, joihin kuuluu geenien monistumisia, translokaatioita ja deleetioita, ja nämä uudelleenjärjestelyt liittyvät usein palindromin läsnäoloon, mikä viittaa mahdolliseen korrelaatioon palindromin ja geenien uudelleenjärjestelyjen välillä. Sitä, miten nämä tapahtumat syntyvät, ei tunneta hyvin. Tiedetään kuitenkin, että tällaiset uudelleenjärjestelyt liittyvät syövän etenemiseen ja ennusteeseen, Rattray sanoi.
Uusi hypoteesi geenien uudelleenjärjestelyistä
Rattrayn mukaan suositussa mallissa, jota alun perin ehdotti yli 60 vuotta sitten tohtori Barbara McClintock, ehdotetaan, että kromosomikatkoksen jälkeen sisaruskromatidit replikoituvat ja fuusioituvat ja synnyttävät kromosomin, jossa on kaksi kromosomikeskipistettä, jotka on yhdistetty toisiinsa dna:ta sisältävällä palindromilla. McClintockin mallissa kahden sentromeerin olemassaolo johtaa uusiin genomin uudelleenjärjestelyihin.
Rattray sanoi kuitenkin, että hänen ryhmänsä ja muut ovat osoittaneet, että ”DNA-palindromit ovat epästabiileja ja voivat itsessään johtaa genomin uudelleenjärjestelyihin, mikä viittaa siihen, että palindromit voivat syntyä sisarkromatidien fuusioitumisen lisäksi myös muiden mekanismien, kuten replikaatiovirheiden, kautta”.”
Ryhmä oletti, että ”syövissä, joissa tapahtuu massiivisia uudelleenjärjestelyjä, solut ovat alttiita palindromien muodostumiselle, ja kun ne ovat muodostuneet, palindromin epävakaus johtaa uusiin uudelleenjärjestelyihin, kuten geenien monistumiseen, translokaatioihin ja deleetioihin”, Rattray sanoi. ”Kaikki geenien uudelleenjärjestelyt ovat mutageenisia, ja sellaiset uudelleenjärjestelyt, jotka edistävät solujen kasvua, kuten syövässä, ovat luonnollisesti valinnan suosiossa.”
Uusi teknologia tunnistaa ja luonnehtii palindromeja
Tutkijat kehittivät teknologian, jonka avulla he voivat tutkia kasvaimia, ja tavoitteena on ymmärtää palindromien muodostumisen todennäköisyyttä näissä kasvaimissa, Rattray sanoi. He toivovat oppivansa, mitkä tapahtumat käynnistävät tällaiset epävakaat muodostumat, ja tämä uusi ymmärrys voisi johtaa uudenlaisiin hoitoihin. Hänen mukaansa ryhmä on jo esimerkiksi todennut, että tietyt hiivasolut, jotka ovat alttiita palindromien muodostumiselle, ovat normaaleja soluja paljon herkempiä säteilylle sekä syövän hoidossa usein käytetyille yhdisteille, kuten sisplatiinille.
”Tällä hetkellä yritän luoda menetelmiä, joilla palindromit voidaan valikoivasti rikastaa muusta solun DNA:sta, mikä mahdollistaa suuremman herkkyyden syöpäsolujen palindromisisällön analysoinnissa”, Rattray sanoi. Aiemmassa menetelmässä tutkijat menettivät risteyssekvenssit, jotka saattaisivat antaa vihjeitä palindromien alkuperästä, ja heidän oli analysoitava ne yksi kerrallaan, hän selitti. ”Olemme nyt osoittaneet, että PacBio-alustalla voi helposti sekvensoida DNA-palindromin läpi”, hän sanoi.
Rattray väitteli tohtoriksi Washingtonin yliopistossa Seattlessa, jossa hän tutki retrovirusten replikaatiota. Columbian yliopistossa suoritetun postdoc-apurahan jälkeen, jossa hän tutki DNA:n rekombinaatiota ja DNA:n kaksoisjuostekatkosten aiheuttamia uudelleenjärjestelyjä hiivassa, hän siirtyi NCI:n palvelukseen Frederickiin työskentelemään GRCBL:n päällikön, tohtori Jeffrey Strathernin laboratorioon.
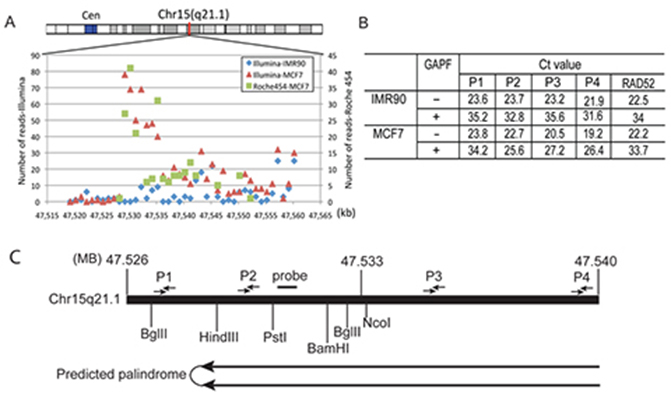
Palindrome-kartoitusstrategia. (A) Lukutiheysjakauma Chr15q21.1:ssä: 47,529,204-47,550,373 alue, joka on esitetty 1 kt:n binneinä. (B) qPCR-analyysi palindromin rikastumisen seuraamiseksi ja Chr15q21.1-palindromin suuntaisuuden määrittämiseksi. Tutkijat laskivat tietyn TaqMan-aloitussarjan alueen köyhtymisen määrän Ct-arvon perusteella ennen ja jälkeen GAPF-protokollan sekä IMR-90- että MCF-7-näytteissä. Kertainen rikastuminen perustuu eri alukesarjojen (P1, P2, P3 ja P4) kertaisen köyhtymisen vertailuun suhteessa genomin yhden kopion sekvenssiin (RAD52). TaqMan-alukesarjojen P1, P2, P3 ja P4 sijainnit on merkitty (C), kartta genomialueesta Chr15: 47,520,000-47,550,000, jossa on rajoituskohdat ja alukkeiden sijainnit. Kuva lähteestä Yang et al., GAP-Seq: a method for identification of DNA palindromes, BMC Genomics 2014, 15:394; doi:10.1186/1471-2164-15-394.
.
