Note de la rédaction : Les articles Platinum Highlight sont des publications remarquables sélectionnées périodiquement par le Dr Craig Reynolds, directeur associé du National Cancer Institute, parmi les publications Platinum les plus récentes.![]()
Lorsqu’Alison Rattray et ses collègues du Gene Regulation and Chromosome Biology Laboratory (GRCBL) ont examiné une cellule de levure mutante qu’ils avaient isolée dans un crible, ils ont remarqué quelque chose d’étrange.
L’ADN présentait un « réarrangement très spécifique, mais bizarre », a-t-elle expliqué. L’arrangement s’est avéré être un palindrome de l’ADN, « ouvrant la porte à l’étude de ces motifs insaisissables de l’ADN », a-t-elle dit.
Scientifique du personnel du GRCBL, Centre de recherche sur le cancer du NCI, Rattray a déclaré que, bien que leur découverte ait eu lieu il y a plusieurs années, leur groupe continue d’étudier la réparation de l’ADN et les réarrangements qui entraînent une réparation anormale « en raison de l’association avec certains cancers ». L’objectif du groupe est de mieux comprendre l’origine de ces réarrangements, a-t-elle ajouté.
Rattray est un contributeur majeur à la recherche rapportée dans BMC Genomics qui décrit la méthode nouvellement développée par le groupe pour le séquençage à l’échelle du génome des palindromes de l’ADN dans une lignée cellulaire cancéreuse.
Qu’est-ce qu’un palindrome d’ADN ?
Une séquence palindromique de nucléotides (qui sont étiquetés A, T, C ou G) se produit lorsque des brins complémentaires d’ADN se lisent de la même façon dans les deux sens, soit à partir de l’extrémité 5-prime, soit à partir de l’extrémité 3-prime. Par exemple, la séquence GGATCC sur un brin d’ADN est considérée comme un palindrome parce que la séquence sur son brin complémentaire est CCTAGG.
En raison de la complémentarité des séquences, les séquences palindromiques peuvent également se replier sur elles-mêmes, formant des boucles en épingle à cheveux ou des cruciformes qui sont extrudés de la double hélice normale, a déclaré Rattray. « Les petites épingles à cheveux ne sont pas problématiques, mais lorsque les palindromes sont longs (plus de 100 paires de bases), ils interfèrent avec les processus cellulaires normaux tels que la transcription et la réplication », a-t-elle expliqué.
Certaines cellules cancéreuses présentent des réarrangements massifs du génome, qui incluent des amplifications, des translocations et des délétions de gènes, et ces réarrangements sont souvent associés à la présence d’un palindrome, ce qui suggère une corrélation possible entre le palindrome et les réarrangements de gènes. La manière dont ces événements se produisent n’est pas bien comprise. Ce que l’on sait, cependant, c’est que ces réarrangements sont associés à la progression et au pronostic du cancer, a déclaré Rattray.
Nouvelle hypothèse pour le réarrangement génomique
Selon Rattray, le modèle privilégié, proposé à l’origine il y a plus de 60 ans par Barbara McClintock, Ph.D., suggère que, suite à une cassure chromosomique, les chromatides sœurs se répliquent et fusionnent, créant un chromosome avec deux centromères joints par un palindrome d’ADN. Dans le modèle de McClintock, le fait d’avoir deux centromères conduit à d’autres réarrangements génomiques.
Toutefois, Rattray a déclaré que son groupe et d’autres ont montré que « les palindromes d’ADN sont instables et peuvent conduire à des réarrangements du génome par eux-mêmes, suggérant en outre que les palindromes pourraient survenir non seulement par la fusion des chromatides sœurs, mais aussi par d’autres mécanismes, tels que des erreurs de réplication. »
Le groupe a émis l’hypothèse que « dans les cancers qui subissent des réarrangements massifs, les cellules sont susceptibles de former des palindromes, et une fois formés, l’instabilité du palindrome conduit à d’autres réarrangements, y compris l’amplification des gènes, les translocations et les délétions », a déclaré Rattray. « Tout réarrangement de gène est mutagène, et les réarrangements qui favorisent la croissance cellulaire, comme dans le cancer, seront naturellement favorisés par la sélection. »
Une nouvelle technologie identifie et caractérise les palindromes
Les chercheurs ont développé une technologie qui leur permettra d’examiner les tumeurs, dans le but de comprendre la probabilité de formation de palindromes dans ces tumeurs, a déclaré Rattray. Ils espèrent apprendre quels événements sont à l’origine de ces formations instables, et cette nouvelle compréhension pourrait conduire à de nouveaux traitements. Par exemple, dit-elle, le groupe a déjà déterminé que certaines cellules de levure qui sont susceptibles de former des palindromes sont beaucoup plus sensibles que les cellules normales aux radiations ainsi qu’aux composés souvent utilisés dans le traitement du cancer, comme le cisplatine.
« Actuellement, j’essaie d’établir des méthodes pour enrichir sélectivement les palindromes du reste de l’ADN cellulaire, ce qui permettra une plus grande sensibilité dans l’analyse du contenu en palindromes des cellules cancéreuses », dit-elle. Dans la méthode précédente, les chercheurs perdaient les séquences de jonction qui pouvaient fournir des indices sur l’origine des palindromes, et devaient les analyser une par une, a-t-elle expliqué. « Nous avons maintenant montré que la plateforme PacBio peut facilement séquencer à travers un palindrome d’ADN », a-t-elle ajouté.
Rattray a obtenu son doctorat à l’Université de Washington, à Seattle, où elle a étudié la réplication rétrovirale. Après un stage postdoctoral à l’Université Columbia, où elle a étudié la recombinaison de l’ADN et les réarrangements induits par les cassures double-brin de l’ADN chez la levure, elle a rejoint le NCI à Frederick, travaillant dans le laboratoire de Jeffrey Strathern, docteur en médecine, chef du GRCBL.
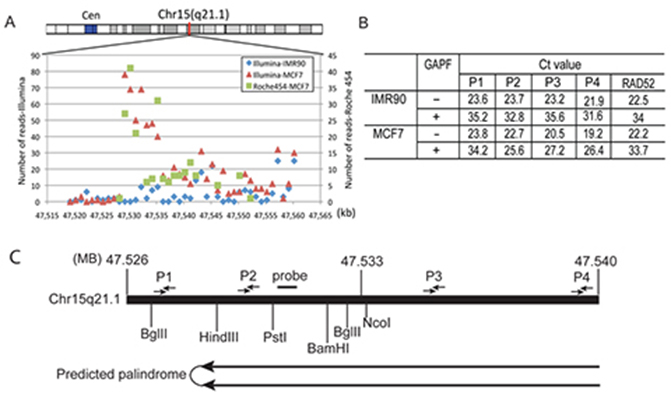
Stratégie de cartographie du palindrome. (A) Distribution de la densité de lecture dans la zone Chr15q21.1 : 47 529 204-47 550 373 région représentée par des bins de 1 kb. (B) Analyse qPCR pour surveiller l’enrichissement du palindrome et déterminer la directionnalité du palindrome de Chr15q21.1. Les chercheurs ont calculé le degré d’appauvrissement d’une région spécifique de l’ensemble d’amorces TaqMan sur la base de la valeur Ct avant et après le protocole GAPF dans les échantillons IMR-90 et MCF-7. L’enrichissement du pli est basé sur la comparaison de l’appauvrissement du pli parmi les différents ensembles d’amorces (P1, P2, P3 et P4) par rapport à une séquence à copie unique dans le génome (RAD52). Les emplacements des ensembles d’amorces TaqMan P1, P2, P3 et P4 sont indiqués dans (C), carte de la région génomique Chr15 : 47 520 000-47 550 000 avec les sites de restriction et les emplacements des amorces. Figure tirée de Yang et al, GAP-Seq : une méthode d’identification des palindromes d’ADN, BMC Genomics 2014, 15:394 ; doi:10.1186/1471-2164-15-394.
.
