
La contribution d’un pont salin à la stabilité globale à l’état plié d’une protéine peut être évaluée par des données thermodynamiques recueillies à partir d’études de mutagenèse et de techniques de résonance magnétique nucléaire. En utilisant une protéine mutée de type pseudo-wild spécifiquement mutée pour empêcher la précipitation à un pH élevé, la contribution du pont salin à l’énergie libre globale de l’état replié de la protéine peut être déterminée en effectuant une mutation ponctuelle, altérant et, par conséquent, brisant le pont salin. Par exemple, un pont salin a été identifié dans le lysozyme T4 entre l’acide aspartique (Asp) au résidu 70 et une histidine (His) au résidu 31 (Figure 3). Une mutagenèse dirigée avec de l’asparagine (Asn) (Figure 4) a été réalisée pour obtenir trois nouveaux mutants : Asp70Asn His31 (Mutant 1), Asp70 His31Asn (Mutant 2), et Asp70Asn His31Asn (Double Mutant).

Une fois les mutants établis, deux méthodes peuvent être employées pour calculer l’énergie libre associée à un pont salin. Une méthode implique l’observation de la température de fusion de la protéine de type sauvage par rapport à celle des trois mutants. La dénaturation peut être suivie par un changement du dichroïsme circulaire. Une réduction de la température de fusion indique une réduction de la stabilité. Ceci est quantifié par une méthode décrite par Becktel et Schellman où la différence d’énergie libre entre les deux est calculée par ΔTΔS. Ce calcul pose quelques problèmes et ne peut être utilisé qu’avec des données très précises. Dans l’exemple du lysozyme T4, le ΔS du type pseudo-doux avait été signalé précédemment à pH 5,5, de sorte que la différence de température à mi-chemin de 11 °C à ce pH multipliée par le ΔS signalé de 360 cal/(mol-K) (1,5 kJ/(mol-K)) donne un changement d’énergie libre d’environ -4 kcal/mol (-17 kJ/mol). Cette valeur correspond à la quantité d’énergie libre contribuant à la stabilité de la protéine par le pont salin.
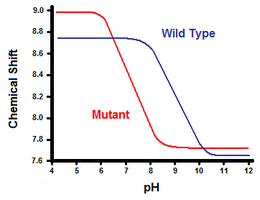
La deuxième méthode utilise la spectroscopie par résonance magnétique nucléaire pour calculer l’énergie libre du pont salin. Un titrage est effectué, tout en enregistrant le déplacement chimique correspondant aux protons du carbone adjacent au groupe carboxylate ou ammonium. Le point médian de la courbe de titrage correspond au pKa, ou au pH où le rapport entre les molécules protonées et déprotonées est de 1:1. En continuant avec l’exemple du lysozyme T4, une courbe de titrage est obtenue par l’observation d’un déplacement du proton C2 de l’histidine 31 (Figure 5). La figure 5 montre le déplacement de la courbe de titrage entre le type sauvage et le mutant dans lequel Asp70 est Asn. Le pont salin formé est entre l’Asp70 déprotoné et l’His31 protoné. Cette interaction provoque le décalage observé dans le pKa de His31. Dans la protéine de type sauvage dépliée, où le pont salin est absent, His31 a un pKa de 6,8 dans des tampons H20 de force ionique modérée. La figure 5 montre un pKa de 9,05 pour la protéine de type sauvage. Cette différence de pKa est soutenue par l’interaction de His31 avec Asp70. Pour maintenir le pont salin, l’His31 tentera de conserver son proton aussi longtemps que possible. Lorsque le pont salin est rompu, comme dans le mutant D70N, le pKa revient à une valeur de 6,9, beaucoup plus proche de celle de l’His31 à l’état déplié.
La différence de pKa peut être quantifiée pour refléter la contribution du pont salin à l’énergie libre. En utilisant l’énergie libre de Gibbs :ΔG = -RT ln(Keq), où R est la constante universelle des gaz, T est la température en kelvins, et Keq est la constante d’une réaction en équilibre. La déprotonation de His31 est une réaction d’équilibre acide avec une Keq particulière connue sous le nom de constante de dissociation acide, Ka : His31-H+ ⇌ His31 + H+. Le pKa est alors relié à Ka par la formule suivante : pKa = -log(Ka). Le calcul de la différence d’énergie libre du mutant et du type sauvage peut maintenant être effectué en utilisant l’équation de l’énergie libre, la définition du pKa, les valeurs de pKa observées et la relation entre logarithmes naturels et logarithmes. Dans l’exemple du lysozyme T4, cette approche a permis de calculer une contribution d’environ 3 kcal/mol à l’énergie libre globale. Une approche similaire peut être adoptée avec l’autre participant du pont salin, tel que l’Asp70 dans l’exemple du lysozyme T4, en surveillant son changement de pKa après la mutation de His31.
Un mot de prudence lors du choix de l’expérience appropriée implique l’emplacement du pont salin dans la protéine. L’environnement joue un rôle important dans l’interaction. À des forces ioniques élevées, le pont salin peut être complètement masqué puisqu’une interaction électrostatique est impliquée. Le pont salin His31-Asp70 du lysozyme T4 était enfoui dans la protéine. L’entropie joue un rôle plus important dans les ponts salins de surface où les résidus qui ont normalement la possibilité de se déplacer sont limités par leur interaction électrostatique et leur liaison hydrogène. Il a été démontré que cela diminue suffisamment l’entropie pour presque effacer la contribution de l’interaction. Les ponts salins de surface peuvent être étudiés de la même manière que les ponts salins enfouis, en utilisant des cycles de double mutant et des titrages RMN. Bien qu’il existe des cas où les ponts salins enfouis contribuent à la stabilité, comme pour toute autre chose, des exceptions existent et les ponts salins enfouis peuvent avoir un effet déstabilisant. De même, les ponts salins de surface, dans certaines conditions, peuvent avoir un effet stabilisant. L’effet stabilisateur ou déstabilisateur doit être évalué au cas par cas et peu de déclarations générales peuvent être faites.
