La mutagenèse dirigée par site (MDS) est une technique utilisée pour muter une ou plusieurs bases au sein d’un plasmide. Cette approche peut modifier la composition des acides aminés, détruire les sites de liaison des facteurs de transcription ou créer des protéines de fusion, pour ne citer que quelques exemples.
Bien que la MDS soit le plus souvent utilisée pour sonder la structure et l’activité biologique des acides nucléiques et des protéines, elle est également utile pour introduire ou supprimer des sites de reconnaissance des enzymes de restriction afin de faciliter le clonage.
En outre, la MDS peut être un outil d’optimisation des codons utile pour remplacer les codons rares dans les vecteurs d’expression afin d’améliorer l’efficacité de l’expression des protéines hétérologues. Cela est parfois nécessaire en raison du biais de codon, un phénomène par lequel les organismes ont une préférence pour certains codons par rapport à d’autres, en fonction de la disponibilité des ARNt dans la cellule. Vous pouvez en savoir plus sur l’utilisation des codons et le biais de codon ici.
Lorsqu’elle est employée pour modifier la séquence codante, la SDM crée des constructions mutantes spécifiques qui manquent de résidus critiques ou de domaines protéiques impliqués dans les modifications post-traductionnelles ou la régulation de la stabilité des protéines. L’élimination de résidus ou de régions critiques dans vos plasmides, comme les sites de phosphorylation ou les domaines critiques, constitue un moyen de démontrer l’importance de certaines caractéristiques des protéines. Par exemple, la modification des sites de phosphorylation, tels que la sérine en alanine non réactive ou en phosphomimétiques tels que l’acide aspartique, fournit des preuves fiables de la phosphorylation spécifique des protéines. Elle permet d’étudier les implications in vitro de telles modifications post-traductionnelles.
Approches traditionnelles de la mutagenèse dirigée par site
PCR inverse
Pour les délétions ou insertions de >50 pb, la PCR inverse est l’approche la plus populaire. La PCR inverse utilise des amorces dos à dos pour amplifier l’ensemble du plasmide, suivi d’une ligature du produit linéaire formant un ADN circulaire. Cette technique convient également pour les insertions ou les délétions plus importantes, par exemple l’élimination d’un domaine de régulation d’une protéine.
Pour les délétions, la zone sélectionnée peut être éliminée en concevant des amorces qui s’hybrident de part et d’autre de la zone de délétion ciblée. L’amplification se déroule vers l’extérieur de cette zone, excluant ainsi cette région du produit de la PCR. Une fois le produit linéaire ligaturé, votre nouvelle construction sera dépourvue de ce domaine de délétion. Ce processus est décrit schématiquement dans la figure 1 ci-dessous.
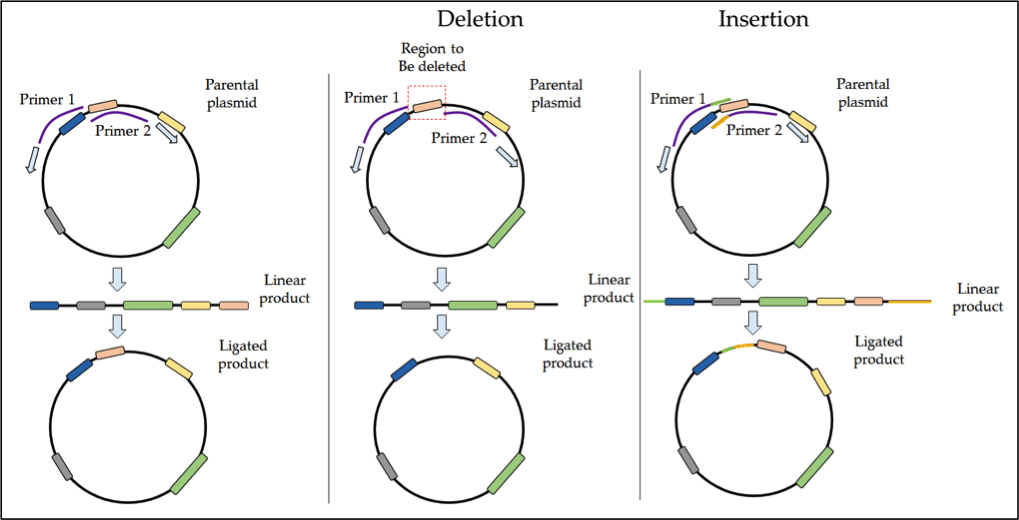
Figure 1. Protocole pour la PCR inverse dans SDM.
Les insertions peuvent être réalisées en utilisant des amorces chevauchantes avec des séquences flanquantes qui contiennent les bases supplémentaires appropriées. Pour plus d’informations sur la PCR inverse, consultez ces ressources :
- Ochman et al. (1998) Applications génétiques d’une réaction en chaîne par polymérase inverse. Génétique. 120:621-623.
- Clifford N. Dominy et David W. Andrews. Mutagenèse dirigée par site par PCR inverse.
SDM en utilisant des amorces modifiées
Cette technique utilise des amorces modifiées pour incorporer de petits changements de paires de bases dans un plasmide, et c’est la méthode de choix pour les mutations spécifiques à un site. Pour commencer, vous aurez besoin de concevoir des amorces. Mais avant de commencer :
- Imprimez une copie d’un tableau de codons d’acides aminés
- Ayez votre séquence de plasmide ou de protéine à portée de main.
- Soyez sûr de savoir où se trouve le codon de départ et dans quel cadre de lecture votre séquence est lue.
Conception des amorces
Visez des amorces SDM d’environ 30 pb de longueur avec votre site muté aussi près du centre que possible. Bien qu’il soit acceptable de faire des amorces un peu plus longues ou plus courtes selon les besoins, il devrait y avoir un minimum de 12 pb de part et d’autre de votre site muté.
Si vous avez besoin d’aide pour la conception d’amorces, http://molbiol-tools.ca/PCR.htm énumère quelques ressources vraiment utiles pour vous mettre sur la voie.
Prendre les bons réactifs
La Taq polymérase ordinaire ne suffit pas quand il s’agit de SDM – vous devez utiliser une enzyme de correction. Il existe une variété de kits de polymérase disponibles dans le commerce qui sont à la hauteur de la tâche, incorporant une gamme de caractéristiques, y compris la haute fidélité (capacité de relecture) et l’activation de démarrage à chaud. Une possibilité est le In-Fusion HD Cloning Plus de Takara – une solution tout en 1 qui comprend une polymérase haute fidélité, un kit de purification PCR, une enzyme de clonage et des cellules compétentes pour la mutagenèse dirigée vers un site.
N’oubliez pas que comme la plupart des réactifs de laboratoire, de nombreuses polymérases ont leurs propres avantages et inconvénients – généralement, les laboratoires ont des raisons historiques de choisir l’une plutôt que l’autre et s’en écartent rarement. Bien qu’il soit raisonnable de s’en tenir à ce qui fonctionne, il n’y a aucun mal à lire la littérature sur d’autres ressources disponibles pour s’assurer que vous avez les meilleurs réactifs pour le travail !
Réaction PCR
Les conditions de PCR utilisées varieront en fonction de votre choix de kit et de polymérase, ainsi que des amorces que vous avez conçues et de la taille du produit. Cependant, l’exemple présenté ci-dessous est un bon point de départ.
| Étape | Temp (°C) | Temps (s) | Cycles |
|---|---|---|---|
| Dénaturation | 94 | 15 | 18 |
| Annealisation | 60 | 30 | 18 |
| Extension | 72 | 20/kb plasmide | 18 |
Tableau 1 : Exemple de programme de cyclage thermique SDM
Maintenir le nombre de cycles bas empêchera les mutations indésirables de se produire. Dix-huit cycles devraient donner une quantité raisonnable de produit muté sans incorporer de mutations indésirables. Notez que le nombre de cycles peut être modifié au cours du dépannage si nécessaire.
Digérer !
Maintenant que vous avez votre produit PCR prêt, n’oubliez pas l’étape critique – la digestion ! Ici, vous digérez l’ADN matrice parental pour vous assurer que vous n’avez que le plasmide muté pour la transformation bactérienne. Les protocoles standard prévoient une incubation de 1 heure à 37°C avec l’endonucléase Dpn1 pour digérer tout l’ADN parental dam-méthylé et hémiméthylé, vous laissant avec votre plasmide muté désiré.
Transformation et séquençage
Transformez votre plasmide dans des cellules compétentes comme vous le feriez avec n’importe quel autre plasmide d’expression, et isolez des colonies uniques pour l’isolement du plasmide.
Maintenant vous devriez avoir un petit volume de votre plasmide désiré. Avant de commencer à expérimenter, envoyez un échantillon pour le séquençage afin de confirmer la présence de votre ou vos modifications souhaitées. Il est tout aussi important de s’assurer de l’absence de toute mutation secondaire indésirable.
Si le séquençage revient avec un résultat positif – félicitations, vous êtes un pro du SDM ! Mais si le séquençage revient avec un résultat négatif – ne vous inquiétez pas ! Vous pouvez toujours réessayer – nos conseils d’experts pour le dépannage du SDM devraient vous remettre sur la bonne voie !
De nos jours, la diminution des coûts de synthèse des oligonucléotides et les progrès de la biologie synthétique signifient que les approches synthétiques gagnent en traction sur la mutagenèse dirigée par site. En outre, l’émergence de la technologie CRISPR/Cas9 a également simplifié l’édition de gènes de sorte que la mutagenèse peut désormais être réalisée in vitro et in vivo en quelques étapes simples. Néanmoins, la MDS continue d’être un pilier de la boîte à outils de la biologie moléculaire et n’est pas prête de disparaître.
Parution initiale en 2016. Mis à jour et republié en 2018.

Cela vous a aidé ? Alors n’hésitez pas à partager avec votre réseau.
.
