部位特異的突然変異誘発(SDM)は、プラスミド内の1つまたは複数の塩基を変異させるために使用される技術です。 このアプローチでは、アミノ酸組成を変更したり、転写因子結合部位を破壊したり、融合タンパク質を作成したりすることができます。
SDMは核酸やタンパク質の構造や生物活性を調べるために最も広く用いられていますが、制限酵素認識部位を導入または除去してクローニングを支援するためにも有用です。 これは、細胞内のtRNAの利用可能性に応じて、生物が特定のコドンを他のコドンよりも優先的に使用する現象であるコドンバイアスのために必要な場合があります。 コドン使用とコドン偏りについて詳しくは、こちらをご覧ください。
コーディング配列を変更するために使用すると、翻訳後修飾やタンパク質の安定性の制御に関わる重要な残基やタンパク質ドメインを欠いた特定の変異体コンストラクトが作成されます。 プラスミド内のリン酸化部位や重要なドメインなどの重要な残基や領域を削除することで、特定のタンパク質機能の重要性を実証する1つの方法となります。 例えば、セリンなどのリン酸化部位を反応性のないアラニンやアスパラギン酸などのリン酸化模倣物質に変更することで、タンパク質の部位特異的リン酸化の信頼性の高い証拠を得ることができる。
Traditional Approaches to Site-Directed Mutagenesis
Inverse PCR
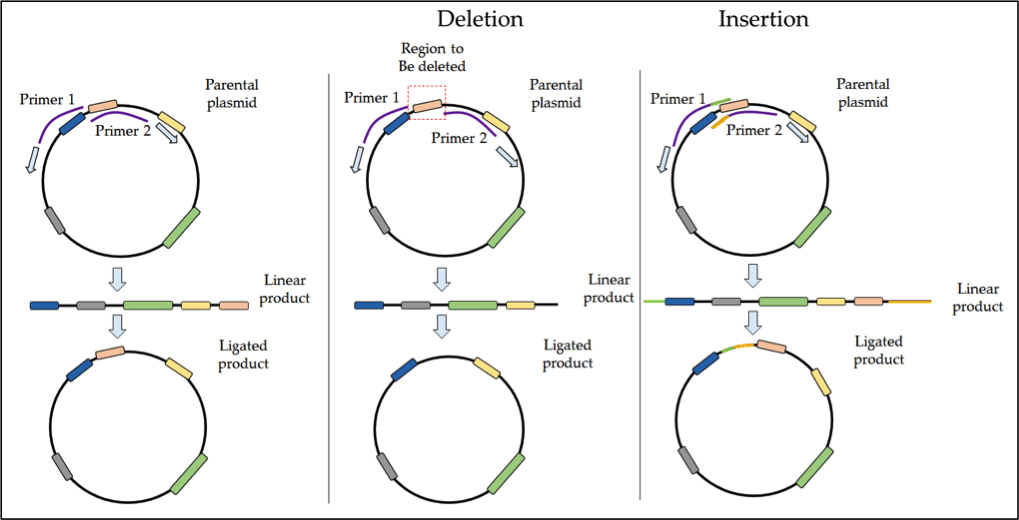
>50 bpの欠損や挿入には、inverse PCRが最も一般的なアプローチである。 Inverse PCRはプラスミド全体を増幅するためにback-to-backのプライマーを使用し、その後、線状産物をライゲーションして円形のDNAを形成させる。 この手法は、例えばタンパク質から制御ドメインを除去するような、より大きな挿入や欠失にも適している。
欠失では、標的とする欠失領域の両側でアニールするプライマーを設計することにより、選択した領域を除去することが可能である。 増幅はこの領域から外側に進み、こうしてPCR産物からこの領域が除かれる。 直鎖状の産物をライゲーションすると、この欠失ドメインを欠いた新しいコンストラクトができあがる。 このプロセスは、以下の図1に概略的に示されています。 SDMにおけるinverse PCRのプロトコル
挿入は、適切な余分な塩基を含むフランキング配列とオーバーラップするプライマーを使用することで達成することができる。 inverse PCRの詳細については、以下の資料を参照してください:
- Ochman et al. (1998) Genetic Applications of an Inverse Polymerase Chain Reaction. Genetics. 120:621-623.
- Clifford N. Dominy and David W. Andrews. インバースPCRによる部位特異的変異誘発.
SDM Using Modified Primers
この技術は、プラスミドに小さな塩基対変化を組み込むために修正プライマーを使用し、部位特異的変異のために選択される方法である。 まず始めに、いくつかのプライマーを設計する必要があります。
- アミノ酸コドン表をプリントアウトする
- プラスミドまたはタンパク質の配列を準備しておく
- 開始コドンがどこにあり、どのリーディングフレームで配列が読まれるかを確認しておく。
プライマーの設計
変異部位をできるだけ中心に寄せて、約30bpの長さのSDMプライマーを目指します。
プライマーの設計で助けが必要な場合、http://molbiol-tools.ca/PCR.htmに本当に役に立つリソースがリストアップされていますので、参考にしてください。 市販のポリメラーゼキットには、ハイフィデリティ(プルーフリーディング能力)やホットスタート活性化など、さまざまな機能が盛り込まれており、この仕事に適したものがある。 このキットには、ハイフィデリティポリメラーゼ、PCR精製キット、クローニング酵素、部位特異的突然変異誘発用のコンピテントセルが含まれています。 うまくいくものにこだわるのは賢明ですが、他の利用可能なリソースの文献を読んで、仕事に最適な試薬があることを確認することに害はありません!
PCR Reaction
使用するPCR条件は、設計したプライマーや産物のサイズと同様に、キットやポリメラーゼの選択によって異なります。 しかし、以下に示す例は良い出発点である。
| Step | Temp (℃) | Time (s) | Cycles |
|---|---|---|---|
| Denature | 94 | 15 | 18 |
| Anneal | 60 | 30 | 18 |
| Extension | 72 | 18 |
表1.アニーリング SDMサーマルサイクリングプログラムの例
サイクル数を少なくすることで、不要な変異の発生を防ぐことができます。 18サイクルであれば、不要な変異を取り込むことなく、適度な量の変異生成物を得ることができるはずです。
Digest It!
さて、PCR産物の準備ができたら、重要なステップである消化を忘れてはいけません! ここでは、親テンプレートDNAを消化して、細菌形質転換のための変異プラスミドのみを確実に得ることができます。 標準的なプロトコルは、エンドヌクレアーゼDpn1と37℃で1時間インキュベートして、すべてのダムメチル化およびヘミメチル化された親DNAを消化し、目的の変異プラスミドを残すことを要求しています。
形質転換と配列決定
他の発現プラスミドと同様に、プラスミドをコンピテントセルに形質転換し、プラスミド単離のためにシングルコロニーを分離します
これで、目的のプラスミドを少量手に入れたはずです。 実験を始める前に、サンプルを送って配列決定を行い、目的の修飾が存在することを確認します。
配列決定が肯定的な結果となった場合、おめでとうございます、あなたはSDMのプロです!
これで、目的のプラスミドを少量持つことができました。 しかし、もしシーケンシングがネガティブな結果であったとしても、気に病む必要はありません。 SDM のトラブルシューティングのエキスパートによるヒントで、軌道に乗るはずです!
最近では、オリゴヌクレオチド合成のコスト低下と合成生物学の進歩により、合成アプローチが部位特異的突然変異誘発よりも人気を集めています。 さらに、CRISPR/Cas9技術の登場により、遺伝子編集が簡略化され、in vitroおよびin vivoでの変異誘発が簡単な手順で行えるようになった。 それでも、SDMは分子生物学のツールボックスの主役であり続け、すぐにはどこにも行きません」
Original published in 2016. 2018年に更新され再出版されました。
これはあなたの助けになりましたか? それでは、あなたのネットワークと共有してください。
