A brief introduction to ChIP-Seq
タンパク質-DNA相互作用は細胞生理学を支えるメカニズムを明らかにするために広く使用されています。 クロマチン免疫沈降(ChIP)アッセイ技術の開発により、そのような機構の研究が可能になった。
ChIP-Seq実験は、まず全細胞をホルムアルデヒドで架橋し、その後超音波処理とDNAの分離を行うことから始まる。 その後、特定のタンパク質に結合する抗体からなるDNA-タンパク質複合体の免疫沈降が行われる。 形成された免疫複合体は沈殿され、精製される。 最後に、DNAの塩基配列を決定し、濃縮された部位の高分解能データを作成する。
臨床的意義と応用
疾患や健康状態におけるエピジェネティックな不均衡には、ヒストン修飾や転写因子の変化が関与している可能性があることが分かっています。 ここで、ChIP-Seq研究は、癌や他の疾患の基礎となる病理学的な分子メカニズムを解明するために使用されています。 また、ChIP-seq解析は、疾患における転写因子の役割の理解にも寄与しています。 実際、いくつかの転写産物は臨床表現型の発現中に変化するようです。
ChIP-Seqパイプラインの概要
ChIP-Seq解析パイプラインは、DNA-タンパク質相互作用プロジェクトの主要コンポーネントで、生データ処理、品質管理解析、参照ゲノムへのアライメント、アライメントしたリードの品質チェック、ピークコール、アノテーション、可視化などの複数のステップから構成されています。 しかし、ChIP-seq実験で高品質の結果を得るためには、考え抜かれた実験デザインを持つことが重要です。 解析を始める前に、サンプル複製、コントロールグループ、シーケンスキット、シーケンスプラットフォームなどのパラメータを考慮することが重要です。
Quality Control
すべてのBasepairレポートは、入力データに潜在するシーケンスの問題や汚染を明らかにするための品質スコアを提供します。 このステップは、DNA-seqおよびRNA-seq解析で実施されるものと類似しています。 ここでは、配列と塩基の品質、GCコンテンツ、シーケンスアダプターの存在、過剰発現配列などが主な評価基準となる。 この種の解析に最もよく使われるプログラムの1つがFastQCです。 さらに、低品質な配列が特定された場合、後でトリミングのステップでそれらを除去することができます。
アライメント
QC測定後、ChIP-Seqリードは参照ゲノムにアライメントされます。 リードマッピングにより、研究者はゲノム内のリード配列の起源を特定することができます。 よく使われるアライメントソフトウェアツールにはBowtieとBWAがあり、BasepairのChIP-seqパイプラインではこの2つが使用されています。
リードカウントフローは、トリミング、アライメント、重複排除プロセスの最後に、使用可能なリードの全体像を把握するのに役立ちます。
Quality check of the aligned reads
次のステップは、アライメントされたデータセットのQC推論である。 マッピングの過程で、PCR増幅やシーケンシングによって生じたリードの重複は、ピークコールやエンリッチメント解析の際にバイアスの原因となる。 BasepairはPicardツールを用いて重複を除去します。 重複を除去したら、アライメントされたリードのNon-Redundant Fraction (NRF)を評価する必要があります。 NRFは、参照ゲノムにマッピングされたユニークリードの数を測定します。
Peak Calling
ピークコールステップは、ゲノム上のタンパク質-DNA相互作用領域が濃縮されていることを検出します。 BasepairのChIP-seqパイプラインは、この解析を行うためにMACS2を使用しています。 MACS2では、フラグメントの推定、局所的なノイズパラメータの同定、ピークの同定の3つの主要なステップに基づいてピークコールが実行されます。 このステップの出力として、ユーザーは、濃縮スコア、-log10pvalue、-log10qvalue、ピーク開始位置などのピーク情報を含む最終テーブルを得ることができます。 このステップでは、調査対象のデータセットと比較するために、コントロールサンプルを使用することを強く推奨します。 5530>
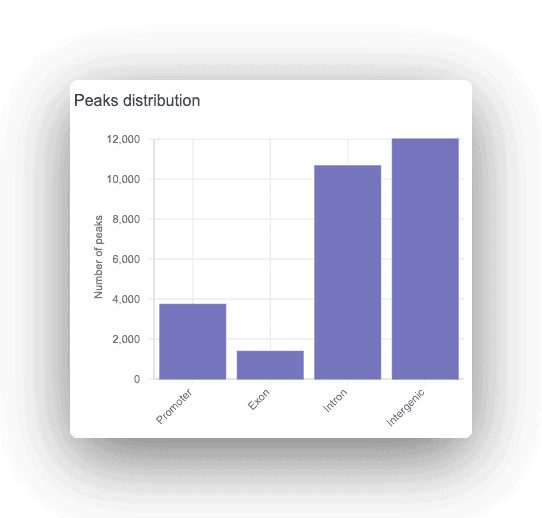
各ピークはプロモーター、イントロン、インタージェニックとして注釈され、対応する遺伝子が表示されます。
結果の概要
ChIP-seqパイプラインはクロマチン状態に関する情報だけでなく、特定の遺伝子または遺伝子座のコンテキストにおける転写因子結合も提供することが可能である。 DNA調節領域におけるヒストン修飾や転写因子の出現は、条件特異的なエピジェネティックシグネチャーを構成する可能性がある。 このように、エピジェネティックな障害は、臨床的な表現型と関連付けることができる。 例えば、クロマチン状態の不均一性は、乳癌の治療抵抗性につながる可能性がある。 これらの細胞は、抑制的なヒストン修飾マーカーを失う傾向があり、がん治療への耐性を促進することが知られている遺伝子の発現がさらに増加します。
ChIP-Seq Analysis Pipelineにおけるピーク、モチーフおよびパスウェイ分析
モチーフ転写因子濃縮の特定は、転写因子が所定の領域で協力または競合しているかを明らかにするために使用されます。 DNAモチーフ領域におけるピークの同定は、実験結果の解釈を向上させることができます。 ピークとモチーフの両方を解析することで、細胞内で何が起こっているのかを知ることができる。 ピークとモチーフの濃縮を統合することで、生物学的影響が考えられるエピゲノムランドスケープを得ることができます。 さらに、パスウェイ解析はパスウェイに含まれるタンパク質を同定するために使用される。 さらに、パスウェイ解析により、パスウェイに含まれるタンパク質を同定し、タンパク質の存在に基づいて調査や結論の策定を行います。
Data Visualization
ChIP-seq パイプラインからの結果データは、ゲノムブラウザを使用して視覚化することができます。 Basepairレポートには、IGV2ゲノムブラウザが組み込まれており、データを操作することができます。 また、ヒートマップ(特定のマークの有無を示すデータ密度に基づく代表的な強度のインフォグラフィックス)を用いて、データを可視化することもできます。 その他、enrichment plot、upSet、coverage plot などのグラフィックがあり、ゲノム上のピーク領域のカバー率を計算し表示します。 BasepairのすべてのChIP-seq解析レポートに搭載されています。
1. Grosselin, K., A. Durand, et al. High-throughput single-cell ChIP-seq identifies heterogeneity of chromatin states in breast cancer.(ハイスループット単一細胞ChIP-seqによる乳癌のクロマチン状態の不均一性の特定)。 Nat Genet, v.51, n.6, Jun, p.1060-1066. 2019.
2.Northrup, D. L. e K. Zhao. ChIP-Seqおよび関連技術の免疫機能研究への応用。 Immunity, v.34, n.6, Jun 24, p.830-42. 2011.
3. Park, S. J., J. H. Kim, et al. Bioconductor Packages に基づく ChIP-Seq Data Analysis Pipeline. Genomics Inform, v.15, n.1, Mar, p.11-18. 2017.
4. Pepke, S., B. Wold, et al. ChIP-seq and RNA-seq studiesのためのComputation. Nat Methods, v.6, n.11 Suppl, Nov, p.S22-32. 2009.
5. 佐藤寿一、川名尚志、他 ChIP-SeqによるNRF1標的遺伝子のパスウェイ解析から、神経変性疾患の病態形成への関与が論理的に推測される。 Gene Regul Syst Bio, v.7, p.139-52. 2013.
