編集部注:Platinum Highlight記事は、国立がん研究所副所長のクレイグ・レイノルズ博士が最近発表したPlatinum Publicationsから定期的に選んだ注目すべき出版物である。![]()
Gene Regulation and Chromosome Biology Laboratory (GRCBL) のAlison Rattray氏と同僚たちは、スクリーニングで分離した変異酵母細胞を調べたところ、奇妙なことに気づきました。
そのDNAには「非常に特殊だが奇妙な転位」が見られたと、彼女は説明します。 その配置は、DNAパリンドロームであることが判明し、「これらのとらえどころのないDNAモチーフを研究する扉を開いたのです」と、彼女は語りました。
NCI Center for Cancer ResearchのGRCBLスタッフ科学者は、彼らの発見は数年前のことですが、彼らのグループはDNA修復と異常修復をもたらす再配置の研究を続けている、と述べ、「特定のがんとの関連性から」述べています。 同グループの目標は、これらの再配列の起源をよりよく理解することであると、彼女は付け加えました。
ラットレイは、BMC Genomicsに報告された研究の主要な貢献者で、癌細胞系におけるDNAパリンドロームのゲノム全体の配列決定について、同グループが新たに開発した方法について述べています。
DNAパリンドロームとは
ヌクレオチド(A、T、C、またはGと表示される)の回文配列は、DNAの相補鎖が5主末または3主末から、両方向に同じ読み方をするときに発生します。 たとえば、DNA の一方の鎖上の配列 GGATCC は、その相補鎖上の配列が CCTAGG であるため、回文とみなされます。
配列の相補性により、回文配列もそれ自身に折り返し、ヘアピン ループや十字形を形成して、通常の二重らせんからはみ出ることがあると、Rattray は述べています。 「
一部の癌細胞は、遺伝子の増幅、転座、欠失を含む大規模なゲノム再配置を示し、これらの再配置はしばしば回文配列の存在と関連しており、回文配列と遺伝子再配置の間に相関関係がある可能性が示唆されます。 これらの事象がどのようにして生じるかはよくわかっていない。 ラットレイ氏によると、バーバラ・マクリントック博士が60年以上前に提唱したモデルが有力で、染色体の切断後、姉妹染色分体が複製・融合し、DNA回文によって2つの動原体が結合した染色体が作られると考えられているとのことである。 しかし、ラットレイ氏は、「DNAパリンドロームは不安定で、それだけでゲノムの再配列を引き起こす可能性があり、さらに、パリンドロームは姉妹染色体の融合だけでなく、複製エラーなど他のメカニズムによっても生じ得ることを示唆している」と述べている。”
研究グループは、「大規模な再配列が起こるがんでは、細胞は回文が形成されやすく、いったん形成されると回文の不安定性が遺伝子の増幅、転座、欠失などさらなる再配列を引き起こす」という仮説を立てています、とRattray氏は述べました。 “どんな遺伝子再配列も変異原性であり、癌のように細胞増殖を促進する再配列は、当然選択によって好まれるでしょう。”
新技術がパリンドロームを特定し特徴づける
研究者達は、これらの腫瘍におけるパリンドローム形成の可能性を理解する目的で、腫瘍を調べることを可能にする技術を開発したと、ラットレイは述べています。 研究者らは、このような不安定な形成がどのような出来事から始まるのかを知りたいと考えており、この新しい理解が、新しい治療法につながる可能性があるとしている。 たとえば、同グループはすでに、パリンドローム形成の影響を受けやすい特定の酵母細胞が、放射線や、シスプラチンなど、がんの治療によく用いられる化合物に対して、正常細胞よりもはるかに感受性が高いことを明らかにしているという。「現在私は、細胞の残りのDNAから選択的にパリンドロームを濃縮する方法を確立しようとしており、これにより、がん細胞のパリンドローム含有量をより敏感に分析することができます」と彼女は述べている。 これまでの方法では、パリンドロームの起源を知る手がかりとなる接合配列が失われ、1つ1つ解析しなければならなかったと、彼女は説明する。 「このたび、PacBioプラットフォームがDNAパリンドロームの配列を容易に決定できることが明らかになりました」と彼女は述べています。 コロンビア大学で博士研究員として、酵母のDNA二本鎖切断によって引き起こされるDNA組み換えと再配列を研究した後、フレデリックのNCIに加わり、GRCBLのチーフであるJeffrey Strathern博士の研究室で働きました。
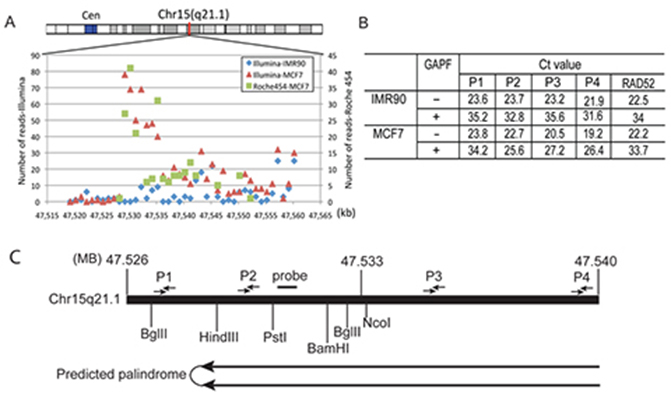
パリンドロームマッピング戦略。 (A)Chr15q21.1におけるリード密度分布。 47,529,204-47,550,373領域における1kbビンとして示した。 (B)パリンドロームの濃縮をモニターし、Chr15q21.1パリンドロームの方向性を決定するためのqPCR解析。 IMR-90とMCF-7の両サンプルで、GAPFプロトコルの前後のCt値に基づいて、特定のTaqManプライマーセット領域の枯渇量を算出した。 ゲノム上の1コピー配列(RAD52)に対する異なるプライマーセット(P1、P2、P3、P4)間の枯渇度を比較することにより、fold enrichmentを算出した。 TaqManプライマーセットP1、P2、P3、P4の位置は、(C)、ゲノム領域Chr15: 47,520,000-47,550,000 の地図と制限部位およびプライマーの位置で示されている。 図はYangら、GAP-Seq: a method for identification of DNA palindromes、BMC Genomics 2014, 15:394; doi:10.1186/1471-2164-15-394.
から引用した。
