Mutagênese Direcionada ao Local (SDM) é uma técnica usada para mutilar uma ou mais bases dentro de um plasmídeo. Esta abordagem pode alterar a composição de aminoácidos, destruir locais de ligação do fator de transcrição, ou criar proteínas de fusão – para citar alguns exemplos.
Embora SDM seja mais amplamente utilizado para sondar a estrutura e atividade biológica dos ácidos nucléicos e proteínas, também é útil para introduzir ou remover locais de reconhecimento de enzimas de restrição para ajudar na clonagem.
Outras vezes, SDM pode ser uma ferramenta útil de otimização de códons para substituir códons raros em vetores de expressão para melhorar a eficiência da expressão de proteínas heterólogas. Isso às vezes é necessário devido ao viés do códão, um fenômeno pelo qual os organismos têm preferência por certos códons em relação a outros, dependendo da disponibilidade de tRNAs na célula. Você pode ler mais sobre o uso do códon e o viés do códon aqui.
Quando empregado para alterar a seqüência de codificação, SDM cria construções mutantes específicas que carecem de resíduos críticos ou domínios protéicos envolvidos em modificações pós-tradução ou regulação da estabilidade protéica. A remoção de resíduos críticos ou regiões em seus plasmídeos, tais como locais de fosforilação ou domínios críticos, fornece uma maneira de demonstrar a importância de certas características protéicas. Por exemplo, a alteração dos sítios de fosforilação, como serina para alanina não reativa ou para fosfomiméticos, como ácido aspártico, produz evidências confiáveis para a fosforilação proteica específica do sítio. Permite o estudo das implicações in vitro de tais modificações pós-tradução.
Adesenvios Tradicionais à Mutagénese Dirigida ao Local
Inversa PCR
Para eliminação ou inserções de >50 bp, a PCR inversa é a abordagem mais popular. A PCR inversa usa primers back-to-back para amplificar todo o plasmídeo, seguido por ligadura do produto linear formando o DNA circular. Esta técnica também é adequada para inserções ou deleções maiores, por exemplo, remover um domínio regulador de uma proteína.
Para deleções, a área selecionada pode ser removida projetando primers que recolem em ambos os lados da zona de deleção alvo. A amplificação procede para fora desta área, excluindo assim esta região do produto PCR. Uma vez que o produto linear é ligado, a sua nova construção estará sem este domínio de eliminação. Este processo é esquematizado na Figura 1 abaixo.
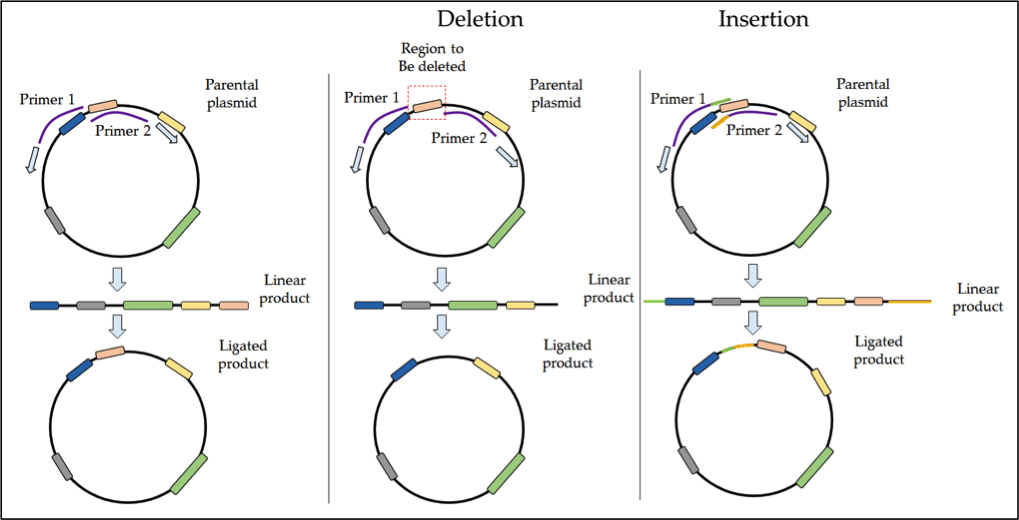
Figure 1. Protocolo para PCR inversa em SDM.
Inserções podem ser obtidas usando primers sobrepostos com seqüências de flanking que contenham as bases extras apropriadas. Para mais informações sobre a PCR inversa verifique estes recursos:
- Ochman et al. (1998) Genetic Applications of an Inverse Polymerase Chain Reaction. Genética. 120:621-623.
- Clifford N. Dominy e David W. Andrews. Mutagénese dirigida ao local por PCR Inversa.
SDM Usando Primers Modificados
Esta técnica usa primers modificados para incorporar pequenas mudanças de par de base em um plasmídeo, e é o método de escolha para mutações específicas do local. Para começar, você precisará projetar alguns primers. Mas antes de começar:
- Imprima uma cópia de uma tabela de aminoácidos de códon
- Deixe a sua sequência de plasmídeos ou proteínas pronta.
- Certifique-se de saber onde está o códon inicial e em que quadro de leitura a sua sequência é lida.
Desenho do Primário
Aponte para primários SDM de aproximadamente 30 bp de comprimento com o seu local mutante o mais próximo possível do centro. Embora seja aceitável fazer primers um pouco mais longos ou mais curtos conforme necessário, deve haver um mínimo de 12 bp de cada lado do seu site mutado.
Se você precisar de ajuda com o design do primer, http://molbiol-tools.ca/PCR.htm lista alguns recursos realmente úteis para colocá-lo em seu caminho.
Pick the Right Reagents
A Taq polimerase ordinária simplesmente não vai cortá-la quando se trata de SDM – você precisa usar uma enzima de revisão. Há uma variedade de kits de polimerase comercialmente disponíveis que estão à altura do trabalho, incorporando uma série de características, incluindo alta fidelidade (capacidade de revisão) e ativação hot start. Uma possibilidade é o In-Fusion HD Cloning Plus da Takara – uma solução tudo em 1 que inclui uma polimerase de alta fidelidade, um kit de purificação PCR, uma enzima de clonagem e células competentes para mutagênese direcionada ao local.
Cuidado que, como a maioria dos reagentes de laboratório, muitas polimerases vêm com seus próprios prós e contras – geralmente os laboratórios têm razões históricas para escolher uma sobre a outra e raramente se desviam disso. Embora seja sensato manter o que funciona, não há mal nenhum em ler a literatura sobre outros recursos disponíveis para se certificar de que tem os melhores reagentes para o trabalho!
ReacçãoPCR
As condições de PCR utilizadas irão variar dependendo da sua escolha do kit e da polimerase, bem como dos iniciadores que concebeu e do tamanho do produto. No entanto, o exemplo mostrado abaixo é um bom ponto de partida.
| Passo | Tempo (°C) | Tempo (s) | Ciclos |
|---|---|---|---|
| Denatura | 94 | 15 | 18 |
| Anneal | 60 | 30 | 18 |
| >Extensão | 72 | 20/kb plasmid | 18 |
Tabela 1: Exemplo de programa de ciclagem térmica SDM
Calcando o número de ciclos baixo, evitará a ocorrência de mutações indesejadas. Dezoito ciclos devem produzir uma quantidade razoável de produto mutado sem incorporar mutações indesejadas. Note que o número de ciclos pode ser alterado durante a resolução de problemas, se necessário.
Digest It!
Agora que tenha o seu produto PCR pronto, não se esqueça do passo crítico – a digestão! Aqui, você digere o DNA do modelo parental para garantir que você só tenha o plasmídeo mutante para a transformação bacteriana. Os protocolos padrão requerem uma incubação de 1 hora a 37°C com endonuclease Dpn1 para digerir todo o ADN parental dam-methylated e hemi-methylated, deixando-o com o plasmídeo mutado desejado.
Transforma e Sequência
Transforma o seu plasmídeo em células competentes tal como faria com qualquer outro plasmídeo de expressão, e isola colónias individuais para isolamento plasmídeo.
Agora deve ter um pequeno volume do plasmídeo desejado. Antes de começar a experimentar, envie uma amostra para sequenciar para confirmar a presença da(s) sua(s) modificação(ões) desejada(s). É igualmente importante assegurar a ausência de quaisquer mutações secundárias indesejadas.
Se a sequenciação regressar com um resultado positivo – parabéns, você é um profissional SDM! Mas se a sequência retornar com um resultado negativo – não se preocupe! Pode sempre tentar novamente – as nossas dicas especializadas para a resolução de problemas SDM deverão colocá-lo novamente no bom caminho!
Agora, actualmente, a diminuição dos custos da síntese de oligonucleótidos e os avanços na biologia sintética significam que as abordagens sintéticas estão a ganhar tracção sobre a mutagénese direccionada para o local. Além disso, o surgimento da tecnologia CRISPR/Cas9 também simplificou a edição de genes de tal forma que a mutagénese pode agora ser realizada in vitro e in vivo em alguns passos simples. No entanto, o SDM continua a ser um pilar fundamental na caixa de ferramentas de biologia molecular, e não vai a lugar algum tão cedo.
Originalmente publicado em 2016. Atualizado e republicado em 2018.

O que isto lhe ajudou? Então por favor compartilhe com sua rede.
