Editor’s note: Platinum Highlight articles are noted publications selected periodically by Dr. Craig Reynolds, associate director, National Cancer Institute, from among the latest published Platinum Publications.![]()
Quando Alison Rattray e colegas do Laboratório de Regulação Genética e Biologia Cromossômica (GRCBL) examinaram uma célula de levedura mutante que tinham isolado em uma tela, eles notaram algo estranho.
O DNA exibia um “rearranjo muito específico, mas estranho”, explicou ela. O arranjo acabou sendo um palíndromo de DNA, “abrindo a porta para o estudo desses motivos esquivos de DNA”, disse ela.
Um cientista do GRCBL, NCI Center for Cancer Research, Rattray disse que, enquanto sua descoberta ocorreu há alguns anos, seu grupo continua a estudar a reparação do DNA e rearranjos que resultam em reparo anormal “por causa da associação com certos cancros”. O objetivo do grupo é entender melhor a origem desses rearranjos, acrescentou ela.
Rattray é um dos principais contribuintes para as pesquisas relatadas na BMC Genomics que descreve o método recém desenvolvido pelo grupo para seqüenciamento de palíndromos de DNA em uma linha de células cancerígenas em todo o genoma.
O que é um palíndromo de ADN?
Ocorre uma sequência palindrómica de nucleótidos (que são rotulados como A, T, C, ou G) quando feixes complementares de ADN lêem o mesmo em ambas as direcções, seja da extremidade de 5-prime ou da extremidade de 3-prime. Por exemplo, a seqüência GGATCC em uma fita de DNA é considerada um palíndromo porque a seqüência em sua fita complementar é CCTAGG.
Por causa da complementaridade da seqüência, as seqüências palíndromas também podem se dobrar para trás, formando loops ou cruciformes que são extrudados da hélice dupla normal, disse Rattray. “Pequenos grampos de cabelo não são problemáticos, mas quando os palíndromos são longos (mais de 100 pares de bases), eles interferem com processos celulares normais, como transcrição e replicação”, explicou ela.
Algumas células cancerígenas exibem rearranjos massivos do genoma, que incluem amplificações, translocações e deleções de genes, e esses rearranjos estão frequentemente associados à presença de um palíndromo, sugerindo uma possível correlação entre o palíndromo e os rearranjos do gene. Como estes eventos surgem não é bem compreendido. O que se sabe, entretanto, é que tais rearranjos estão associados à progressão e prognóstico do câncer, disse Rattray.
Nova Hipótese para Rearranjo Genômico
De acordo com Rattray, o modelo preferido, proposto originalmente há mais de 60 anos por Barbara McClintock, Ph.D., sugere que, após uma quebra cromossômica, cromatídeos irmãos se replicam e se fundem, criando um cromossomo com dois centrômeros unidos por um palíndromo de DNA. No modelo McClintock, ter dois centrômeros leva a rearranjos genômicos adicionais.
No entanto, Rattray disse que seu grupo e outros mostraram que “palíndromos de DNA são instáveis e podem levar a rearranjos genômicos por si mesmos, sugerindo ainda que palíndromos poderiam surgir não apenas pela fusão cromatídica irmã, mas também por outros mecanismos, tais como erros de replicação.”
O grupo formulou a hipótese de que “em cânceres que sofrem rearranjos maciços, as células são suscetíveis à formação de palíndromos, e uma vez formados, a instabilidade do palíndromo leva a rearranjos adicionais, incluindo amplificação, translocações e deleções de genes”, disse Rattray. “Qualquer rearranjo genético é mutagênico, e rearranjos que promovem o crescimento celular, como no câncer, serão naturalmente favorecidos pela seleção”.
Nova Tecnologia Identifica e Caracteriza Palíndromos
Os pesquisadores desenvolveram tecnologia que lhes permitirá examinar tumores, com o objetivo de compreender a probabilidade de formação de palíndromos nesses tumores, disse Rattray. Eles esperam saber que eventos iniciam tais formações instáveis, e este novo entendimento pode levar a novos tratamentos. Por exemplo, disse ela, o grupo já determinou que certas células de levedura que são susceptíveis à formação de palíndromos são muito mais sensíveis que as células normais à radiação, bem como aos compostos frequentemente utilizados no tratamento do câncer, como a cisplatina.
“Atualmente, estou tentando estabelecer métodos para enriquecer seletivamente os palíndromos do resto do DNA celular, o que permitirá uma maior sensibilidade na análise do conteúdo palíndromo das células cancerosas”, disse ela. No método anterior, os pesquisadores perderam as sequências de junção que poderiam fornecer pistas sobre a origem dos palíndromos, e tiveram que analisá-los um a um, explicou ela. “Mostramos agora que a plataforma PacBio pode rapidamente seqüenciar através de um palíndromo de DNA”, disse ela.
Rattray recebeu seu doutorado na Universidade de Washington, em Seattle, onde estudou a replicação retroviral. Após uma bolsa de pós-doutorado na Universidade de Columbia, onde estudou recombinação de DNA e rearranjos induzidos por quebra dupla de DNA em levedura, ela se juntou ao NCI em Frederick, trabalhando no laboratório de Jeffrey Strathern, Ph.D., chefe do GRCBL.
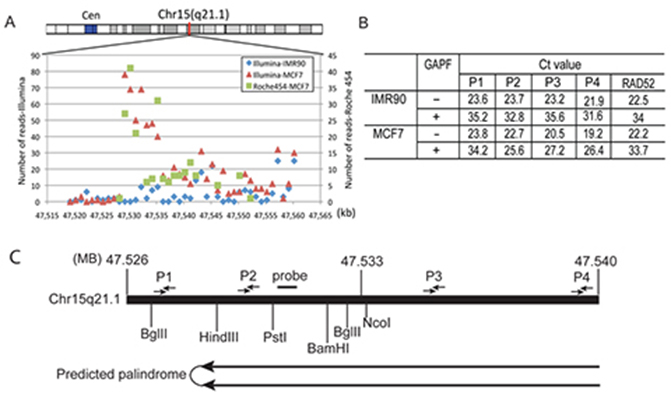
Estratégia de mapeamento do palíndromo. (A) Distribuição da densidade de leitura no Chr15q21.1: 47.529.204-47.550.373 região mostrada como silos de 1 kb. (B) Análise qPCR para monitorar o enriquecimento do palíndromo e determinar a direcionalidade do palíndromo Chr15q21.1. Os pesquisadores calcularam a quantidade de depleção de uma região específica do conjunto de primers TaqMan com base no valor Ct antes e depois do protocolo GAPF tanto nas amostras IMR-90 como MCF-7. O enriquecimento da dobra baseia-se na comparação do esgotamento da dobra entre diferentes conjuntos de iniciadores (P1, P2, P3 e P4) em relação a uma única sequência de cópias no genoma (RAD52). As localizações dos conjuntos de primers TaqMan P1, P2, P3 e P4 estão indicadas em (C), mapa da região genómica Chr15: 47.520.000-47.550.000 com locais de restrição e localizações dos primers. Figura de Yang et al., GAP-Seq: um método para identificação de palíndromos de DNA, BMC Genomics 2014, 15:394; doi:10.1186/1471-2164-15-394.
