
Il contributo di un ponte salino alla stabilità complessiva allo stato ripiegato di una proteina può essere valutato attraverso dati termodinamici raccolti da studi di mutagenesi e tecniche di risonanza magnetica nucleare. Usando una proteina pseudo-wild-type mutata specificamente mutata per prevenire la precipitazione a pH elevato, il contributo del ponte salino all’energia libera complessiva dello stato ripiegato della proteina può essere determinato eseguendo una mutazione puntiforme, alterando e, di conseguenza, rompendo il ponte salino. Per esempio, è stato identificato un ponte salino nel lisozima T4 tra l’acido aspartico (Asp) al residuo 70 e un’istidina (His) al residuo 31 (Figura 3). La mutagenesi sito-diretta con asparagina (Asn) (Figura 4) è stata fatta ottenendo tre nuovi mutanti: Asp70Asn His31 (mutante 1), Asp70 His31Asn (mutante 2), e Asp70Asn His31Asn (doppio mutante).

Una volta stabiliti i mutanti, due metodi possono essere impiegati per calcolare l’energia libera associata a un ponte di sale. Un metodo prevede l’osservazione della temperatura di fusione della proteina wild-type rispetto a quella dei tre mutanti. La denaturazione può essere monitorata attraverso un cambiamento nel dicroismo circolare. Una riduzione della temperatura di fusione indica una riduzione della stabilità. Questo è quantificato attraverso un metodo descritto da Becktel e Schellman dove la differenza di energia libera tra i due è calcolata attraverso ΔTΔS. Ci sono alcuni problemi con questo calcolo e può essere usato solo con dati molto accurati. Nell’esempio del lisozima T4, il ΔS del tipo pseudo-wild era stato precedentemente riportato a pH 5,5, quindi la differenza di temperatura media di 11 °C a questo pH moltiplicata per il ΔS riportato di 360 cal/(mol-K) (1,5 kJ/(mol-K)) produce un cambiamento di energia libera di circa -4 kcal/mol (-17 kJ/mol). Questo valore corrisponde alla quantità di energia libera che il ponte salino contribuisce alla stabilità della proteina.
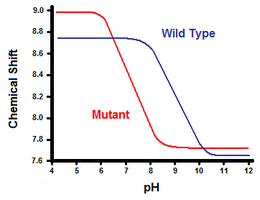
Il secondo metodo utilizza la spettroscopia di risonanza magnetica nucleare per calcolare l’energia libera del ponte salino. Viene eseguita una titolazione, mentre si registra lo spostamento chimico corrispondente ai protoni del carbonio adiacente al gruppo carbossilato o ammonio. Il punto medio della curva di titolazione corrisponde al pKa, o al pH in cui il rapporto tra molecole protonate e deprotonate è 1:1. Continuando con l’esempio del lisozima T4, si ottiene una curva di titolazione attraverso l’osservazione di uno spostamento del protone C2 dell’istidina 31 (Figura 5). La Figura 5 mostra lo spostamento della curva di titolazione tra il wild-type e il mutante in cui Asp70 è Asn. Il ponte salino formato è tra l’Asp70 deprotonato e l’His31 protonato. Questa interazione causa lo spostamento visto nel pKa di His31. Nella proteina wild-type dispiegata, dove il ponte salino è assente, è stato riportato che His31 ha un pKa di 6.8 in tamponi H20 di moderata forza ionica. La figura 5 mostra un pKa del wild-type di 9,05. Questa differenza di pKa è supportata dall’interazione di His31 con Asp70. Per mantenere il ponte salino, His31 cercherà di mantenere il suo protone il più a lungo possibile. Quando il ponte salino è interrotto, come nel mutante D70N, il pKa si sposta di nuovo ad un valore di 6.9, molto più vicino a quello di His31 nello stato dispiegato.
La differenza nel pKa può essere quantificata per riflettere il contributo del ponte salino all’energia libera. Usando l’energia libera di Gibbs: ΔG = -RT ln(Keq), dove R è la costante universale dei gas, T è la temperatura in kelvin, e Keq è la costante di equilibrio di una reazione in equilibrio. La deprotonazione di His31 è una reazione di equilibrio acida con una Keq speciale conosciuta come la costante di dissociazione acida, Ka: His31-H+ ⇌ His31 + H+. Il pKa è quindi legato a Ka dalla seguente formula: pKa = -log(Ka). Il calcolo della differenza di energia libera del mutante e del wild-type può ora essere fatto usando l’equazione di energia libera, la definizione di pKa, i valori di pKa osservati e la relazione tra logaritmi naturali e logaritmi. Nell’esempio del lisozima T4, questo approccio ha prodotto un contributo calcolato di circa 3 kcal/mol all’energia libera complessiva. Un approccio simile può essere preso con l’altro partecipante al ponte salino, come Asp70 nell’esempio del lisozima T4, monitorando il suo spostamento in pKa dopo la mutazione di His31.
Una parola di cautela nella scelta dell’esperimento appropriato riguarda la posizione del ponte salino all’interno della proteina. L’ambiente gioca un ruolo importante nell’interazione. Ad alte forze ioniche, il ponte salino può essere completamente mascherato poiché è coinvolta un’interazione elettrostatica. Il ponte salino His31-Asp70 nel lisozima T4 era sepolto all’interno della proteina. L’entropia gioca un ruolo maggiore nei ponti salini superficiali dove i residui che normalmente hanno la capacità di muoversi sono costretti dalla loro interazione elettrostatica e dal legame a idrogeno. È stato dimostrato che questo diminuisce l’entropia abbastanza da cancellare quasi il contributo dell’interazione. I ponti salini di superficie possono essere studiati in modo simile a quello dei ponti salini sepolti, impiegando cicli di doppi mutanti e titolazioni NMR. Anche se esistono casi in cui i ponti salini sepolti contribuiscono alla stabilità, come qualsiasi altra cosa, esistono eccezioni e i ponti salini sepolti possono mostrare un effetto destabilizzante. Inoltre, i ponti salini superficiali, in certe condizioni, possono mostrare un effetto stabilizzante. L’effetto stabilizzante o destabilizzante deve essere valutato caso per caso e poche dichiarazioni generali possono essere fatte.
