La mutagenesi diretta al sito (SDM) è una tecnica usata per mutare una o più basi in un plasmide. Questo approccio può cambiare la composizione degli aminoacidi, distruggere i siti di legame dei fattori di trascrizione, o creare proteine di fusione, per citare alcuni esempi.
Anche se la SDM è più ampiamente utilizzata per sondare la struttura e l’attività biologica degli acidi nucleici e delle proteine, è anche utile per introdurre o rimuovere i siti di riconoscimento degli enzimi di restrizione per aiutare il clonaggio.
Inoltre, la SDM può essere un utile strumento di ottimizzazione dei codoni per sostituire i codoni rari nei vettori di espressione per migliorare l’efficienza di espressione delle proteine eterologhe. Questo è talvolta necessario a causa del bias dei codoni, un fenomeno per cui gli organismi hanno una preferenza per certi codoni rispetto ad altri, a seconda della disponibilità di tRNA nella cellula. Puoi leggere di più sull’uso dei codoni e sul codon bias qui.
Quando viene impiegato per cambiare la sequenza codificante, l’SDM crea specifici costrutti mutanti che mancano di residui critici o domini proteici coinvolti nelle modifiche post-traslazionali o nella regolazione della stabilità delle proteine. La rimozione di residui critici o regioni nei vostri plasmidi, come i siti di fosforilazione o i domini critici, fornisce un modo per dimostrare l’importanza di certe caratteristiche della proteina. Per esempio, alterando i siti di fosforilazione, come la serina in alanina non reattiva o in fosfomimetici come l’acido aspartico, si ottengono prove affidabili della fosforilazione proteica sito-specifica. Permette lo studio delle implicazioni in vitro di tali modifiche post-traslazionali.
Approcci tradizionali alla mutagenesi diretta al sito
PCDR inversa
Per delezioni o inserzioni di >50 bp, la PCR inversa è l’approccio più popolare. La PCR inversa utilizza primer back-to-back per amplificare l’intero plasmide, seguito dalla legatura del prodotto lineare che forma il DNA circolare. Questa tecnica è adatta anche per inserzioni o delezioni più grandi, ad esempio la rimozione di un dominio di regolazione da una proteina.
Per le delezioni, l’area selezionata può essere rimossa progettando primer che si annebbiano su entrambi i lati della zona di delezione mirata. L’amplificazione procede verso l’esterno di questa zona, escludendo così questa regione dal prodotto della PCR. Una volta che il prodotto lineare è legato, il vostro nuovo costrutto sarà privo di questo dominio di delezione. Questo processo è delineato schematicamente nella figura 1 qui sotto.
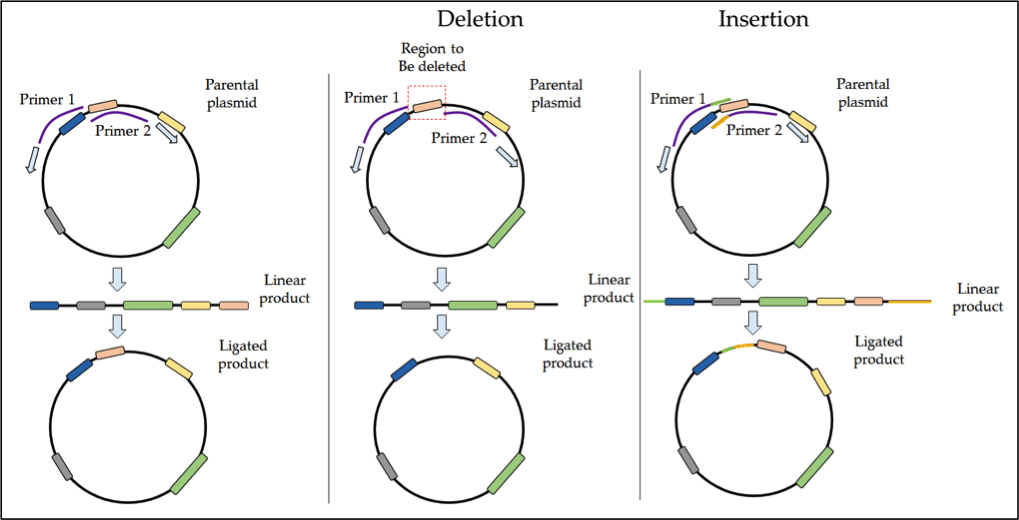
Figura 1. Protocollo per la PCR inversa in SDM.
Le inserzioni possono essere ottenute utilizzando primer sovrapposti con sequenze fiancheggiatrici che contengono le basi extra appropriate. Per ulteriori informazioni sulla PCR inversa controlla queste risorse:
- Ochman et al. (1998) Genetic Applications of an Inverse Polymerase Chain Reaction. Genetica. 120:621-623.
- Clifford N. Dominy e David W. Andrews. Mutagenesi sito-diretta tramite PCR inversa.
SDM Using Modified Primers
Questa tecnica usa primer modificati per incorporare piccoli cambiamenti di coppie di basi in un plasmide, ed è il metodo di scelta per mutazioni sito-specifiche. Per iniziare, avrete bisogno di progettare alcuni primer. Ma prima di iniziare:
- Stampate una copia di una tabella dei codoni degli aminoacidi
- Avete la vostra sequenza di plasmidi o proteine a portata di mano.
- Assicuratevi di sapere dove si trova il codone iniziale e in quale quadro di lettura viene letta la vostra sequenza.
Progettazione di primer
Mirate per primer SDM di circa 30 bp di lunghezza con il vostro sito mutato il più vicino possibile al centro. Mentre è accettabile fare primer un po’ più lunghi o più corti, come richiesto, ci dovrebbe essere un minimo di 12 bp su entrambi i lati del tuo sito mutato.
Se hai bisogno di aiuto con la progettazione dei primer, http://molbiol-tools.ca/PCR.htm elenca alcune risorse davvero utili per metterti sulla tua strada.
Scegli i reagenti giusti
La Taq polimerasi ordinaria non è sufficiente quando si tratta di SDM – è necessario utilizzare un enzima di correzione. Ci sono una varietà di kit di polimerasi disponibili in commercio che sono all’altezza del lavoro, incorporando una serie di caratteristiche tra cui l’alta fedeltà (capacità di correzione) e l’attivazione a caldo. Una possibilità è In-Fusion HD Cloning Plus di Takara – una soluzione all in 1 che include una polimerasi ad alta fedeltà, un kit di purificazione PCR, un enzima di clonazione e cellule competenti per la mutagenesi sito-diretta.
Tieni presente che, come la maggior parte dei reagenti da laboratorio, molte polimerasi hanno i loro pro e contro – generalmente i laboratori avranno ragioni storiche per scegliere una piuttosto che l’altra e raramente si allontaneranno da questo. Mentre attenersi a ciò che funziona è ragionevole, non c’è niente di male nel leggere la letteratura su altre risorse disponibili per assicurarsi di avere i reagenti migliori per il lavoro!
Reazione PCR
Le condizioni PCR utilizzate varieranno a seconda della scelta del kit e della polimerasi, così come i primer che hai progettato e la dimensione del prodotto. Tuttavia, l’esempio mostrato di seguito è un buon punto di partenza.
| Step | Temp (°C) | Tempo (s) | Cicli |
|---|---|---|---|
| Denatura | 94 | 15 | 18 |
| Anneal | 60 | 30 | 18 |
| Extension | 72 | 20/kb plasmide | 18 |
Tabella 1: Esempio di programma di cicli termici SDM
Mantenendo basso il numero di cicli si eviteranno mutazioni indesiderate. Diciotto cicli dovrebbero produrre una quantità ragionevole di prodotto mutato senza incorporare mutazioni indesiderate. Si noti che il numero di cicli può essere modificato durante la risoluzione dei problemi, se necessario.
Digest It!
Ora che hai il tuo prodotto PCR pronto, non dimenticare il passo critico – digestione! Qui, digerisci il DNA del template parentale per assicurarti di avere solo il plasmide mutato per la trasformazione batterica. I protocolli standard richiedono un’incubazione di 1 ora a 37°C con l’endonucleasi Dpn1 per digerire tutto il DNA parentale dam-metilato ed emi-metilato, lasciandoti con il plasmide mutato desiderato.
Trasforma e sequenzia
Trasforma il tuo plasmide in cellule competenti come faresti con qualsiasi altro plasmide di espressione, e isola singole colonie per l’isolamento del plasmide.
Ora dovresti avere un piccolo volume del tuo plasmide desiderato. Prima di iniziare a sperimentare, inviate un campione per il sequenziamento per confermare la presenza della modifica desiderata. È altrettanto importante assicurare l’assenza di qualsiasi mutazione secondaria indesiderata.
Se il sequenziamento ritorna con un risultato positivo – congratulazioni, sei un professionista SDM! Ma se il sequenziamento ritorna con un risultato negativo – non preoccuparti! Puoi sempre provare di nuovo – i nostri suggerimenti di esperti per la risoluzione dei problemi SDM dovrebbero farti tornare in pista!
Al giorno d’oggi, la diminuzione dei costi della sintesi di oligonucleotidi e i progressi nella biologia sintetica significano che gli approcci sintetici stanno guadagnando trazione rispetto alla mutagenesi sito-diretta. Inoltre, l’emergere della tecnologia CRISPR/Cas9 ha anche semplificato l’editing genico in modo che la mutagenesi può ora essere eseguita in vitro e in vivo in pochi semplici passaggi. Ciononostante, la SDM continua ad essere un pilastro nella cassetta degli attrezzi della biologia molecolare, e non andrà da nessuna parte tanto presto.
Originariamente pubblicato nel 2016. Aggiornato e ripubblicato nel 2018.

Ti è stato utile? Allora per favore condividi con la tua rete.
