Mededeling van de auteur: Platinum Highlight artikelen zijn opmerkelijke publicaties die periodiek door Dr. Craig Reynolds, associate director, National Cancer Institute, worden geselecteerd uit de meest recent gepubliceerde Platinum Publications.![]()
Toen Alison Rattray en collega’s in het Gene Regulation and Chromosome Biology Laboratory (GRCBL) een gemuteerde gistcel onderzochten die ze in een screening hadden geïsoleerd, viel hun iets vreemds op.
Het DNA vertoonde een “zeer specifieke, maar vreemde, herschikking”, legde ze uit. De opstelling bleek een DNA-palindroom te zijn, “de deur openend naar het bestuderen van deze ongrijpbare DNA-motieven,” zei ze.
Een stafwetenschapper in GRCBL, NCI Center for Cancer Research, zei Rattray dat, hoewel hun ontdekking een aantal jaren geleden plaatsvond, hun groep doorgaat met het bestuderen van DNA-reparatie en herschikkingen die resulteren in abnormale reparatie “vanwege de associatie met bepaalde kankers.” Het doel van de groep is om de oorsprong van deze herschikkingen beter te begrijpen, voegde ze eraan toe.
Rattray levert een belangrijke bijdrage aan onderzoek gerapporteerd in BMC Genomics dat de nieuw ontwikkelde methode van de groep beschrijft voor genoombrede sequencing van DNA-palindromen in een kankercellijn.
Wat is een DNA palindroom?
Een palindromische sequentie van nucleotiden (die worden gelabeld met A, T, C, of G) treedt op wanneer complementaire strengen van DNA in beide richtingen hetzelfde lezen, hetzij vanaf het 5-priem einde of het 3-priem einde. Bijvoorbeeld, de sequentie GGATCC op één streng van DNA wordt beschouwd als een palindroom omdat de sequentie op zijn complementaire streng CCTAGG is.
Omwille van de complementariteit van de sequentie, kunnen palindromische sequenties ook op zichzelf terugvouwen, waarbij ze haarspeldlussen of kruisvormen vormen die uit de normale dubbele helix worden geëxtrudeerd, zei Rattray. “Kleine haarspelden zijn niet problematisch, maar wanneer palindromen lang zijn (meer dan 100 basenparen), interfereren ze met normale cellulaire processen zoals transcriptie en replicatie,” legde ze uit.
Sommige kankercellen vertonen massale genoomherschikkingen, die genamplificaties, translocaties en deleties omvatten, en deze herschikkingen worden vaak geassocieerd met de aanwezigheid van een palindroom, wat een mogelijke correlatie suggereert tussen het palindroom en de genherschikkingen. Hoe deze gebeurtenissen ontstaan is niet goed begrepen. Wat wel bekend is, is dat dergelijke herschikkingen samenhangen met de progressie en prognose van de kanker, aldus Rattray.
Nieuwe hypothese voor genomische herschikking
Volgens Rattray suggereert het favoriete model, meer dan 60 jaar geleden voorgesteld door Barbara McClintock, Ph.D., dat, na een chromosoombreuk, zusterchromatiden repliceren en fuseren, waardoor een chromosoom ontstaat met twee centromeren die verbonden zijn door een DNA palindroom. In het McClintock-model leidt het hebben van twee centromeren tot verdere genomische herschikkingen.
Rattray zei echter dat haar groep en anderen hebben aangetoond dat “DNA-palindromen instabiel zijn en op zichzelf tot genoomherschikkingen kunnen leiden, wat verder suggereert dat palindromen niet alleen door zusterchromatidefusie zouden kunnen ontstaan, maar ook door andere mechanismen, zoals replicatiefouten.”
De groep veronderstelde dat “in kankers die massale herschikkingen ondergaan, de cellen vatbaar zijn voor palindroomvorming, en eenmaal gevormd, leidt de instabiliteit van het palindroom tot verdere herschikkingen, waaronder genamplificatie, translocaties en deleties,” zei Rattray. “Elke genherschikking is mutageen, en herschikkingen die celgroei bevorderen, zoals bij kanker, zullen natuurlijk door selectie worden bevoordeeld.”
Nieuwe technologie identificeert en karakteriseert palindromen
De onderzoekers ontwikkelden technologie waarmee ze tumoren kunnen onderzoeken, met het doel de waarschijnlijkheid van palindroomvorming in deze tumoren te begrijpen, zei Rattray. Ze hopen te leren welke gebeurtenissen dergelijke onstabiele formaties initiëren, en dit nieuwe begrip zou kunnen leiden tot nieuwe behandelingen. Ze zei bijvoorbeeld dat de groep al heeft vastgesteld dat bepaalde gistcellen die gevoelig zijn voor palindroomvorming veel gevoeliger zijn dan normale cellen voor straling en voor verbindingen die vaak worden gebruikt bij de behandeling van kanker, zoals cisplatine.
“Momenteel probeer ik methoden vast te stellen om palindromen selectief te verrijken van de rest van het cellulaire DNA, waardoor een grotere gevoeligheid mogelijk wordt bij de analyse van het palindroomgehalte van kankercellen,” zei ze. Bij de vorige methode verloren de onderzoekers de verbindingsreeksen die aanwijzingen zouden kunnen geven over de oorsprong van de palindromen, en moesten ze die een voor een analyseren, legde ze uit. “We hebben nu laten zien dat het PacBio-platform gemakkelijk een sequentie kan maken door een DNA-palindroom”, zei ze.
Rattray promoveerde aan de Universiteit van Washington, in Seattle, waar ze retrovirale replicatie bestudeerde. Na een postdoctorale studie aan de Columbia University, waar zij DNA-recombinatie en herschikkingen als gevolg van DNA-dubbelstrengsbreuken in gist bestudeerde, kwam zij in dienst van het NCI in Frederick, waar zij werkte in het laboratorium van Jeffrey Strathern, Ph.D., hoofd, GRCBL.
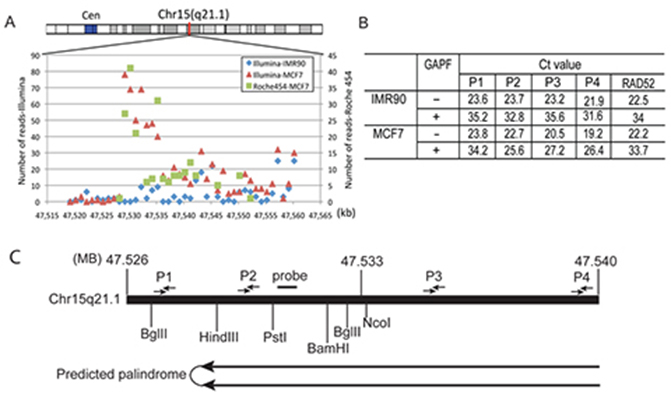
Palindroomkarteringsstrategie. (A) Verdeling van de leesdichtheid in Chr15q21.1: 47.529.204-47.550.373 regio weergegeven als 1-kb bins. (B) qPCR analyse om te controleren op palindroom verrijking en de directionaliteit van het Chr15q21.1 palindroom te bepalen. Onderzoekers berekenden de mate van uitputting van een specifieke TaqMan primerset regio op basis van Ct-waarde voor en na GAPF protocol in zowel IMR-90 en MCF-7 monsters. De vouwverrijking is gebaseerd op de vergelijking van de vouw depletie tussen verschillende primersets (P1, P2, P3, en P4) ten opzichte van een enkele kopie sequentie in het genoom (RAD52). De locaties van de TaqMan-primersets P1, P2, P3 en P4 zijn aangegeven in (C), kaart van de genomische regio Chr15: 47.520.000-47.550.000 met restrictieplaatsen en primerlocaties. Figuur uit Yang et al., GAP-Seq: een methode voor identificatie van DNA palindromen, BMC Genomics 2014, 15:394; doi:10.1186/1471-2164-15-394.
