Distanțierii interni transcriși (ITS) sunt regiuni din cadrul transcriptului ribosomal care sunt excizate și degradate în timpul maturării. Secvențele lor prezintă, în general, mai multe variații decât secvența ribosomală, ceea ce le face populare pentru analiza filogenetică și/sau identificarea speciilor și tulpinilor. În special în cazul ciupercilor, aceasta este o tehnică populară de identificare, deoarece identificarea bazată pe caracteristicile morfologice este laborioasă și, adesea, nu conduce la un rezultat corect. Regiunea utilizată este, de obicei, o combinație de secvențe ITS și de secvențe ribozomale și cel mai adesea constă într-o secvență (parțială) de ARNr 18S (ARNr 16S pentru procariote), o regiune transcrisă intern (ITS1), întreaga secvență de ARNr 5.8s, o regiune transcrisă intern (ITS2) și o secvență (parțială) de ARNr 28s. Multe secvențe ITS sunt disponibile în baze de date publice, cum ar fi NCBI și EBI.
Fluxul de lucru pentru tipărirea ITS în software-ul BIONUMERICS
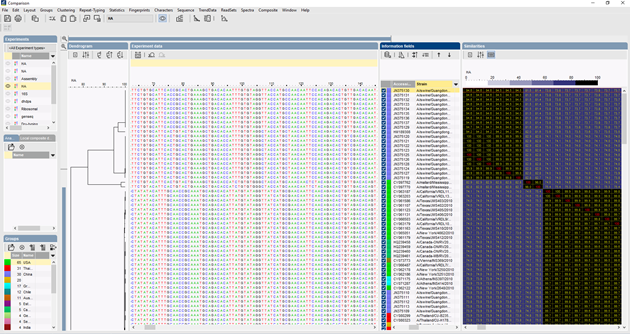
În BIONUMERICS, secvențele ITS pot fi importate în lot din fișiere de urmărire folosind plugin-ul de asamblare a secvențelor în lot. Secvențele de referință pot fi importate din baze de date publice și/sau din fișiere fasta sau GenBank.
Se pot utiliza mai mulți algoritmi pentru a realiza o aliniere multiplă a secvențelor, Needleman-Wunsch, Wilbur-Lipman și propriul nostru algoritm proprietar elaborat la Applied Maths. Este posibilă adaptarea manuală a alinierii. Pe baza alinierii multiple, se poate calcula o grupare care să reflecte relațiile filogenetice dintre secvențele analizate.
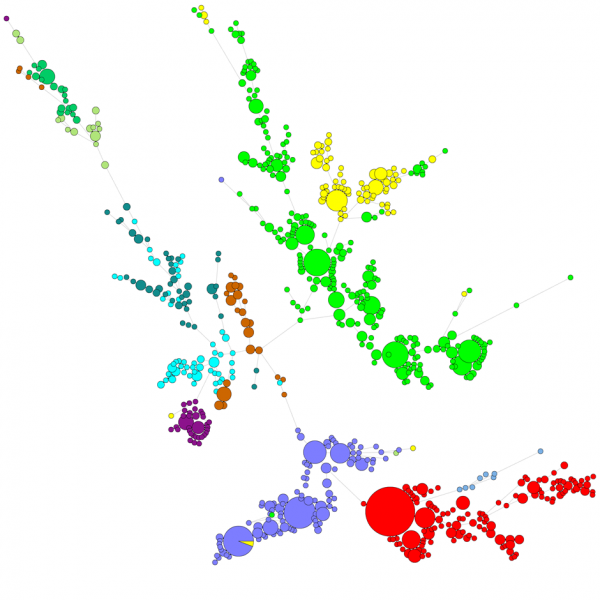
Când se compară un număr mare de secvențe, o dendrogramă UPGMA sau o dendrogramă de îmbinare a vecinilor nu oferă o imagine de ansamblu foarte bună a datelor. În aceste cazuri, un arbore neînrădăcinat oferă o soluție mai bună, în BIONUMERICS este posibil să se calculeze arbori cu întindere minimă, arbori de maximă verosimilitate și arbori de maximă parcimonie pornind de la datele secvenței. Se pot defini diferite grupuri pentru date, iar aceste grupuri sunt vizualizate în arbori, oferind o imagine de ansamblu frumoasă a relațiilor filogenetice, a grupărilor prezente în baza de date și, de asemenea, a valorilor aberante. Figura de mai jos prezintă un arbore minim de cuprindere bazat pe secvențe ITS, grupurile reprezintă diferite genuri.
Secvența ITS poate fi, de asemenea, utilizată pentru a identifica organisme necunoscute în raport cu o bază de date. Cu ajutorul modulului Classifiers and Identification (Clasificatori și identificare), utilizatorul poate crea proiecte de identificare care conțin tulpini identificate și le poate utiliza pentru a identifica o tulpină necunoscută, un scor va fi atribuit pe baza similitudinii, împreună cu fiabilitatea acestui scor. Utilizatorul are control deplin asupra parametrilor de comparație și a valorilor limită pentru identificare.
.
