Internal Transcribed Spacers (ITS) är en region inom det ribosomala transkriptet som skärs av och bryts ned under mognaden. Deras sekvenser uppvisar i allmänhet större variation än den ribosomala sekvensen, vilket gör dem populära för fylogenetisk analys och/eller identifiering av arter och stammar. Särskilt när det gäller svampar är detta en populär teknik för identifiering eftersom identifiering baserad på morfologiska egenskaper är arbetskrävande och ofta inte leder till ett korrekt resultat. Den region som används är vanligtvis en kombination av ITS- och ribosomsekvenser och består oftast av en (partiell) 18S rRNA-sekvens (16S rRNA för prokaryoter), en internt transkriberad region (ITS1), hela sekvensen av 5,8s rRNA, en internt transkriberad region (ITS2) och en (partiell) sekvens av 28s rRNA. Många ITS-sekvenser finns tillgängliga i offentliga databaser som NCBI och EBI.
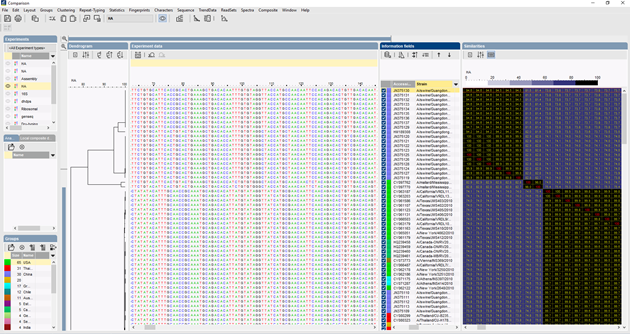
Arbetsflöde för ITS-typning i programvaran BIONUMERICS
I BIONUMERICS kan ITS-sekvenserna importeras i batch från spårfiler med hjälp av insticksmodulen för batch-sekvensmontering. Referenssekvenser kan importeras från offentliga databaser och/eller från fasta- eller GenBank-filer.
Flera algoritmer kan användas för att utföra en multipel anpassning av sekvenserna, Needleman-Wunsch, Wilbur-Lipman och vår egen proprietära algoritm som utarbetats på Applied Maths. Manuell anpassning av anpassningen är möjlig. Baserat på den multipla anpassningen kan ett kluster beräknas för att återspegla de fylogenetiska relationerna mellan de analyserade sekvenserna.
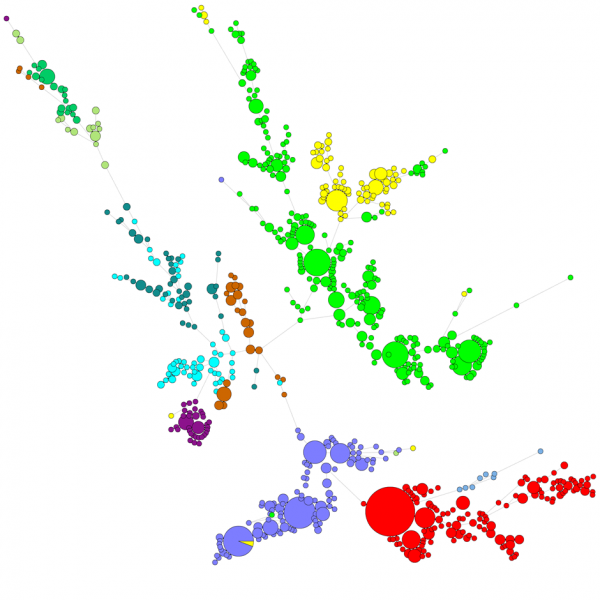
När man jämför ett stort antal sekvenser ger ett UPGMA- eller neighbor-joining-dendrogram inte en särskilt bra överblick över data. I dessa fall ger ett orotat träd en bättre lösning, i BIONUMERICS är det möjligt att beräkna minimal spanning trees, maximum likelihood trees och maximum parsimony trees med utgångspunkt från sekvensdata. Olika grupper kan definieras för data och dessa grupper visualiseras i träden, vilket ger en bra översikt över de fylogenetiska relationerna, de kluster som finns i databasen och även outliers. Figuren nedan visar ett minimalt spännande träd baserat på ITS-sekvenser, grupperna representerar olika släkten.
ITS-sekvensen kan också användas för att identifiera okända organismer mot en databas. Med modulen Classifiers and Identification kan användaren skapa identifieringsprojekt som innehåller identifierade stammar och använda dessa för att identifiera en okänd stam, en poäng kommer att tilldelas baserat på likheten tillsammans med tillförlitligheten av denna poäng. Användaren har full kontroll över parametrarna för jämförelse och gränsvärdena för identifiering.
