内部転写スペーサー(ITS)はリボソーム転写物中の成熟期に切除、分解された領域であります。 その配列は一般にリボソーム配列よりも多くの変異を示すため、系統解析や種や株の同定によく利用される。 特に真菌類では、形態学的特徴に基づく同定は手間がかかり、正しい結果につながらないことが多いため、同定によく用いられる手法である。 使用する領域は、通常ITSとリボソーム配列の組み合わせで、18S rRNA(原核生物では16S rRNA)の(部分)配列、内部転写領域(ITS1)、5.8s rRNAの全配列、内部転写領域(ITS2)、28s rRNA(部分)配列からなるものがほとんどである。 BIONUMERICSにおけるITSタイピングのワークフロー
BIONUMERICSでは、batch sequence assembly pluginを用いて、トレースファイルからITS配列を一括でインポートすることができる。 参照配列は公開データベースやfastaファイル、GenBankファイルから取り込むことができます。
配列の多重アラインメントには、Needleman-Wunsch、Wilbur-Lipman、およびApplied Mathsで開発した独自のアルゴリズムが使用可能です。 手動でアライメントを調整することも可能です。 2473> 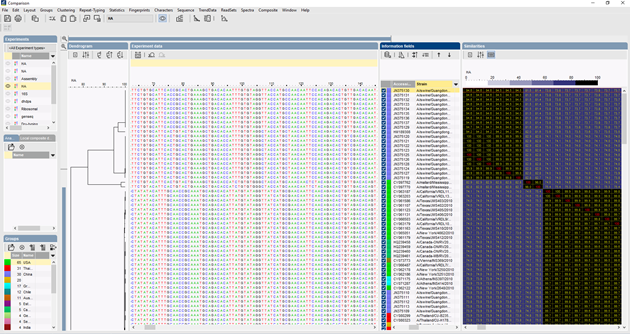
大量の配列を比較する場合、UPGMAやneighbor-joining dendrogramではデータの概要がよくわからない。 BIONUMERICSでは、配列データから最小スパンツリー、最尤ツリー、最大パーシモンツリーを計算することが可能です。 データに対して様々なグループを定義することができ、これらのグループは樹形で可視化されるため、系統関係、データベース内に存在するクラスター、および外れ値の概要を把握することができます。 下図は ITS 配列に基づく最小スパンツリーで、グループは異なる属を表しています。
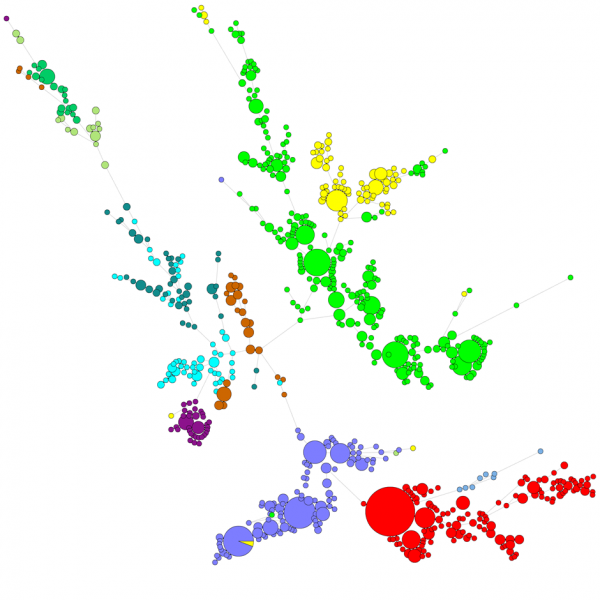
ITS 配列は、データベースに対して未知の生物を識別するために使用することも可能です。 Classifiers and Identification モジュールにより、ユーザーは同定された株を含む同定プロジェクトを作成し、未知の株を同定するためにこれらを使用することができ、このスコアの信頼性とともに類似性に基づいてスコアが割り当てられる。 ユーザーは、比較のためのパラメータと同定のためのカットオフ値を完全に制御することができます
。
